如何分析KEGG Brite数据库,相信很多没有经验的人对此束手无策,为此本文总结了问题出现的原因和解决方法,通过这篇文章希望你能解决这个问题。
KEGG被称为京都基因组百科全书,是一个综合性的数据库。对于如此庞大的数据库,肯定需要对数据进行分门别类的整理。除了将各种数据拆分到不同的子数据库中之外,KEGG还对所有的数据进行了更加细致的功能分类,这些功能分类的信息就存储在brite 数据库中。
birte 主要包含以下五大类别的分类信息:
genes and protein
compounds and reactions
drugs
diseases
organisms and cells
在brite数据库中,以文件的形式存储分类信息。包含两种格式的文件:
table 格式,比如对药物的分类
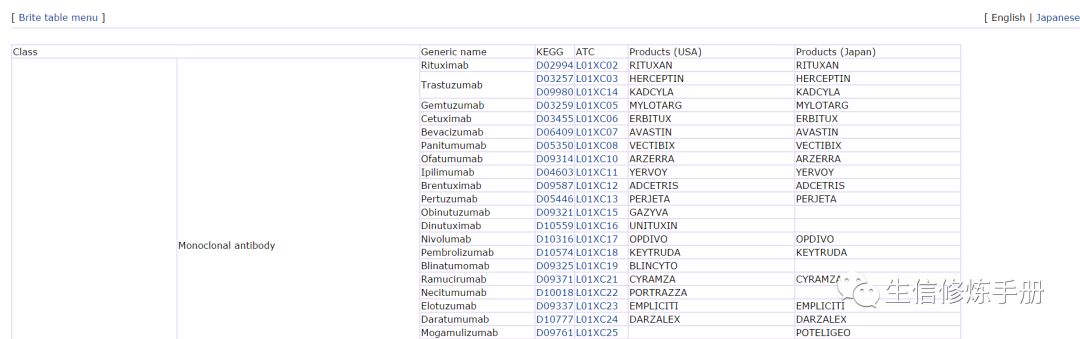
htext 文件,比如kegg orthology 的分类
http://www.kegg.jp/kegg-bin/get_htext?ko00000.keg
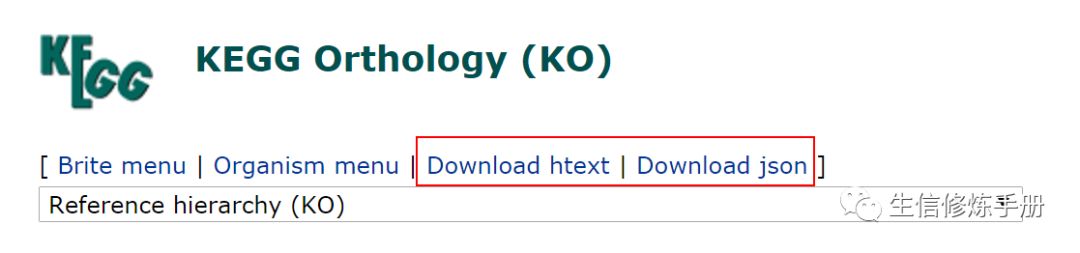
提供了两种格式的文件用于下载,htext 对应的后缀为 keg, json 对应json。
json 格式是网络数据传说的新标准,主要用于程序解析;`keg 文件是纯文本文件,可以用文本编辑器打开。
以所有ko的分类文件 ko00000.keg 文件为例:
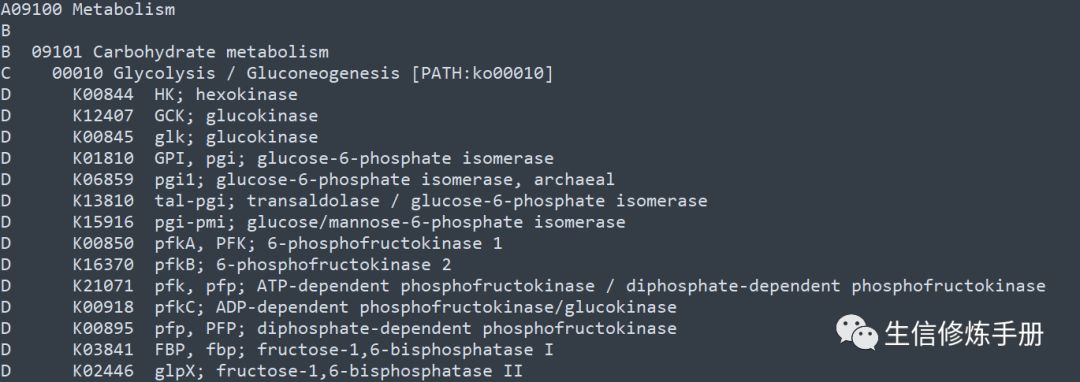
分类层级按照字母顺序排列,示例文件中A 为第一级分类,B, C, D 依次为第二级。
我们可以直观的看到 K00844 属于Glycolysis / Gluconeogenesis 这个分类,对应的更上一级的分类为Carbohydrate metabolism,再上一级为 Metabolism。
keg 文件格式还是非常容易理解的,但是使用起来不够直观,当我们想要查询某个KO的具体分类时,如果和这个KO处于同一分类的节点太多时,需要往上翻阅很多行,才能找到对应的分类;有时一不小心就翻过了,就会搞错。
当然可以通过程序格式化这个文件,比如将这个文件变成如下的格式:
| KO | Name | C | B | A |
|---|---|---|---|---|
| K00844 | HK… | Glycolysis… | Carbo..bolism | Metabolism |
这样方便查看条目的详细分类信息;
对于没有编程基础的人来说,kegg 提供了keggHier 程序,专门用于查看brite中的分类信息。软件是用java 开发的,提供了图形界面,简单易用;
下载地址 :
http://www.kegg.jp/kegg/download/kegtools.html
使用方法
双加批处理文件启动
从菜单栏点击File按钮,选择导入kegg网站上的数据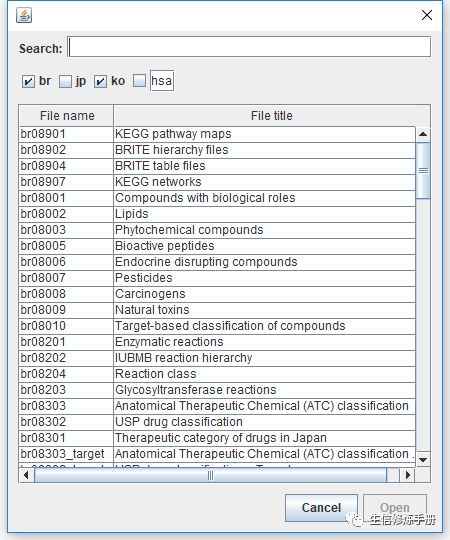
这里选择第一个kegg pathway map 的分类结构,进行查看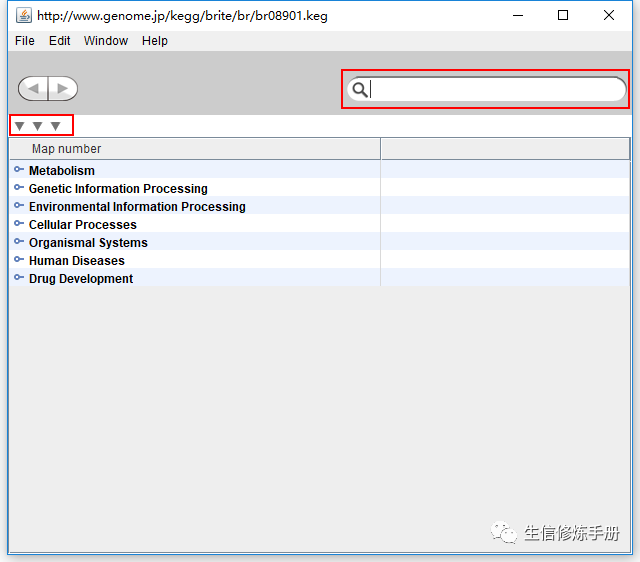
向下的三角形表示展开的意思,这里有3个,说明pathway 共有3层分类,鼠标可以点击任意一条记录,可以展开详细信息;
右上角的搜索框可以搜索,通过搜索框可以快速查找你感兴趣的记录
brite 是存储分类信息的数据库,提供了包含pathway, ko, module, drug, disease,organism 等所有记录的分类;
分类信息通过文件进行距离,有keg 和table两种格式;
通过KEGGHier工具,可以方便的浏览 KEGG 分类系统;
看完上述内容,你们掌握如何分析KEGG Brite数据库的方法了吗?如果还想学到更多技能或想了解更多相关内容,欢迎关注亿速云行业资讯频道,感谢各位的阅读!
亿速云「云数据库 MySQL」免部署即开即用,比自行安装部署数据库高出1倍以上的性能,双节点冗余防止单节点故障,数据自动定期备份随时恢复。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4580290/blog/4626699