怎么使用FitHiC评估染色质交互作用的显著性,相信很多没有经验的人对此束手无策,为此本文总结了问题出现的原因和解决方法,通过这篇文章希望你能解决这个问题。
通过Hi-C技术可以得到全基因组范围内的染色质交互信息, 在不同的分辨率下,首先得到bin之间的交互矩阵contact matrix, 通过热图的形式来展示该交互矩阵,即得到了contact map。在完整的contac matrix的基础上,可以进行A/B隔室,拓扑结构域,染色质环等不同层级空间结构单元的分析。
正是由于覆盖了全基因组范围内的染色质交互信息,才使得hi-C技术可以站在全基因组的高度对不同层级的空间结构进行挖掘,这个是hi-c技术独有的优势。作为3C技术的升级版,hi-c也是可以直接研究某些染色质之间的交互作用的,只不过由于测序和序列比对等系统误差的存在,在交互矩阵中还是有部分信息是不可靠的,为了通过hi-c技术来直接分析某些染色质之间的互作,科学家发明了很多的算法,对交互矩阵中的信息进行评估,通过打分等形式来提取限制性的交互信息,而FitHic就是其中最常用的一款软件。
该软件最初采用python进行开发,后来为了使用方便,将相关功能重写并封装成了一个R包。
该软件的原理示意如下
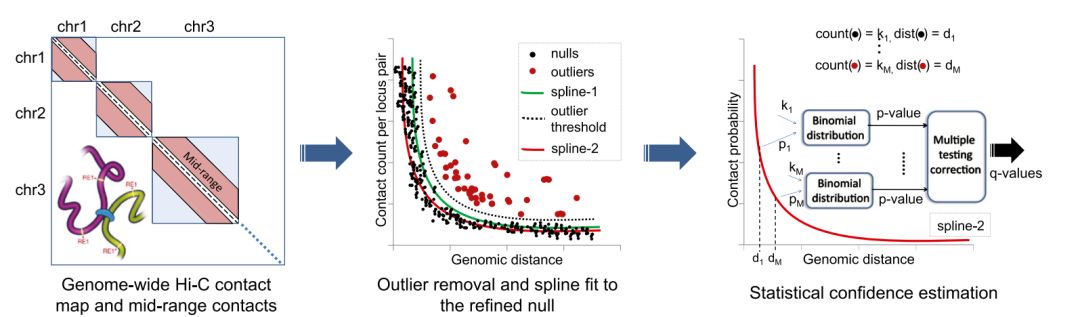
从原始的交互矩阵中,根据事先定义的距离阈值提取出mid-range,即中等距离的同一个染色质bin之间的交互作用。在文章中指出,对于酵母,中等距离的范围为10kb到25kb, 对于人和小鼠,中等距离的范围为50kb到10Mb, 这里的距离为两个bin之间的线性距离。
根据提取出的mid-range交互信息,首先构建基因组线性距离与交互频率的模型,即图中的spline-1, 在该模型的基础上制定过滤的阈值,即虚线代表的outlier-threshold, 然后提出离群值数据,对应图中的红色原点。对于剩下的数据再次进行拟合,得到spline2。 然后在二项分布的基础上计算每个交互作用的pvalue,再进行多种假设检验的校正,得到qvalue。
该软件的用法简单,只不过需要对原始的交互矩阵进行格式化。一个经典的交互矩阵如下所示
Bin1 Bin2 Bin3 Bin4 Bin5 Bin67.85957 4.80329 11.4766 9.57416 4.5288 8.550228.61621 4.98956 2.35654 5.69483 11.1187 10.13224.06803 4.07801 7.98047 2.59144 6.3851 7.743064.52869 2.70624 8.94544 4.29185 8.29491 8.38257
每一行和每一列都代表一个bin, 数字代表两个bin之间的交互频率。在此文件的基础上,经过如下两步即可得到显著性评估的结果。
该软件至少需要准备两个输入文件,第一个文件为bin对应的染色质区域,称之为fragsfile, 内容示意如下
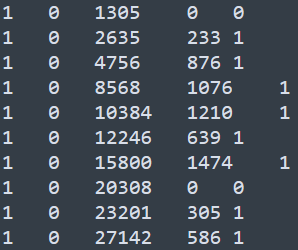
\t分隔的5列,其中第二列和第五列的信息没有作用,用0或者1填充就可以了,第一列表示bin所在的染色体,第三列代表bin的中心位置, 第三列代表与该bin存在交互的频率总和,即交互矩阵中对应列或者行的总和。
第二个文件为bin之间交互频率的信息,称之为intersfile, 内容示意如下
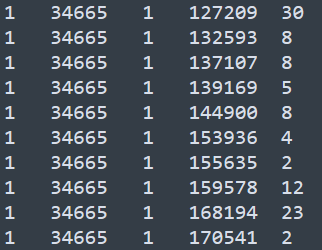
\t分隔的5列,前两列代表第一个bin的染色质名称和中心位置,第三列和第四列代表第二个bin的染色质名称和中心位置,第五列代表两个bin之间的交互频率。
准备好输入文件之后,就可以运行了,基本用法如下
FitHiC( fragsfile, intersfile, outdir, libname = "test_project", distUpThres = 250000, distLowThres = 10000, visual = TRUE)指定两个输入文件和输出结果的目录,libname指定输出文件的前缀,distUpThres和distLowThres指定距离的上下阈值,以此阈值来筛选得到mid-range。
在输出结果中所有文件分成了pass1和pass2两个部分,每个部分有对应的以下4张图
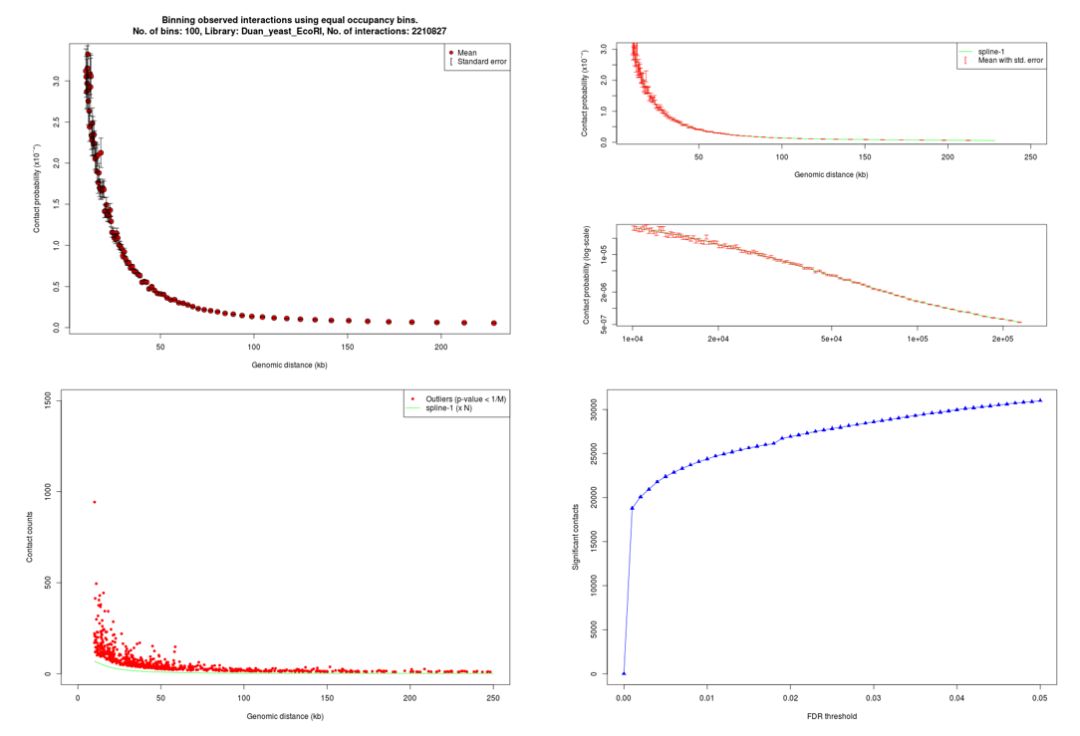
第一张图表示基于mid-range的交互信息得到的基因组线性距离与交互概率的分布,第二张图表示拟合得到的分布,第三张图表示拟合模型筛选得到的离群值,第四张图表示不同FDR阈值筛选的显著交互作用的分布。
最终得到的显著性评估结果可以从后缀为pass2.significances.txt.gz的文件中得到,该文件内容示意如下
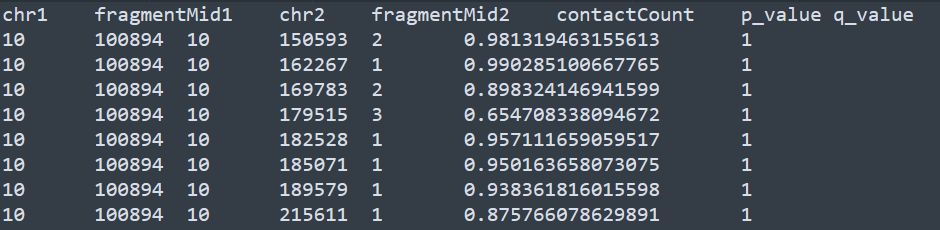
通过最后一列的qvaue作为阈值,去筛选得到显著性的染色质互作。
看完上述内容,你们掌握怎么使用FitHiC评估染色质交互作用的显著性的方法了吗?如果还想学到更多技能或想了解更多相关内容,欢迎关注亿速云行业资讯频道,感谢各位的阅读!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4580290/blog/4570565