这篇文章主要为大家展示了“Oncomine肿瘤芯片数据库的示例分析”,内容简而易懂,条理清晰,希望能够帮助大家解决疑惑,下面让小编带领大家一起研究并学习一下“Oncomine肿瘤芯片数据库的示例分析”这篇文章吧。
肿瘤作为人类健康的头号杀手,其研究的重要性不言而喻。随着芯片和NGS技术的发展,发表了很多的肿瘤相关数据。然而这些数据来自不同的组织和团队,由于缺乏统一的数据管理和组织,这些数据在发表之后就没有再利用了,为了提高数据利用率,促进肿瘤研究的发展,Oncomine的开发团队收集了各种来源的肿瘤相关的芯片数据,用标准化的分析流程处理这些数据,数据分析的结果通过web服务查询和可视化,对应的文章链接如下
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1635162/
数据库的网址如下
https://www.oncomine.org/resource/main.html
该网站只对科研工作者免费开放,需要学术邮箱注册对应的账号。该数据库中主要收录了以下两种类型的肿瘤芯片数据
mRNA expression
DNA copy number
每批芯片数据用dataset表示,所有芯片数据对应的肿瘤类型和样本数示意如下
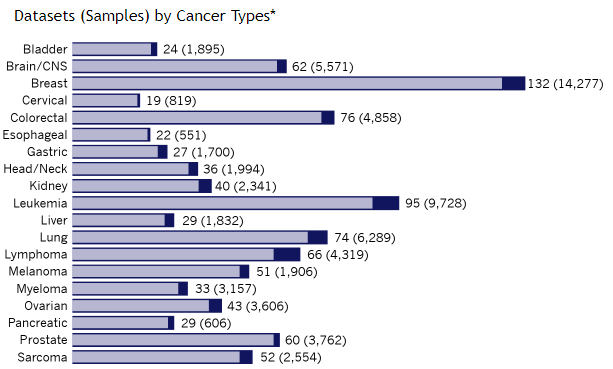
对于芯片数据,主要进行了以下几种类型的分析
coexpression analyses
differential expression analyses
outlier analyses
首页的面板分成了3个部分,示意如下
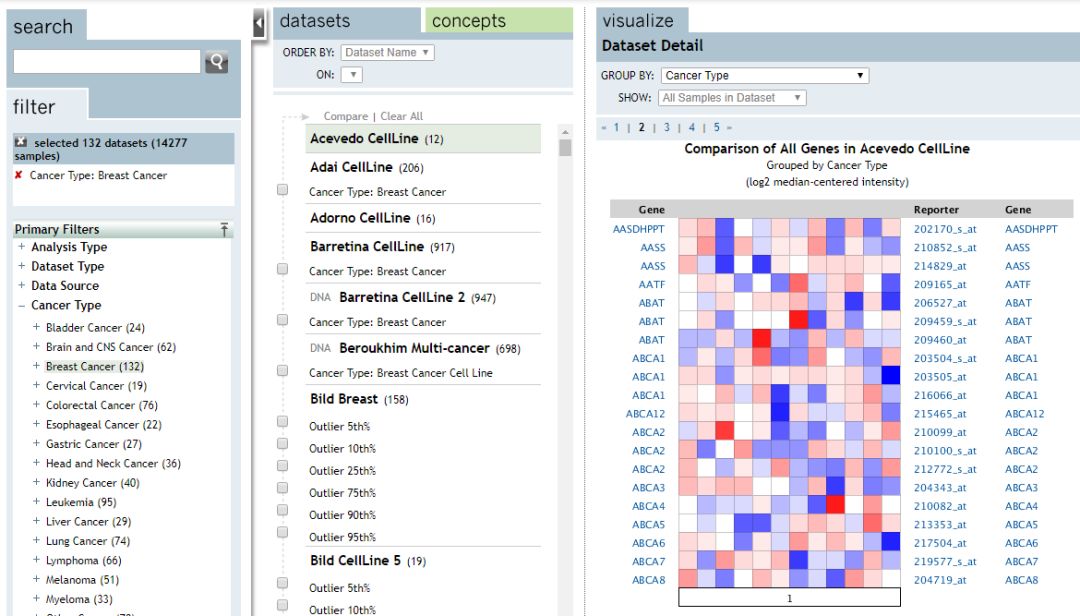
最左侧的部分用于对数据进行检索或者筛选,中间的面板用于展示所有的数据集,最右侧的面板用于显示数据集的详细结果。最基本的展现形式是热图,示意如下
在search框中指定一个感兴趣的基因,然后可以查看在特定数据集中与该基因存在共表达的基因结果,示意如下
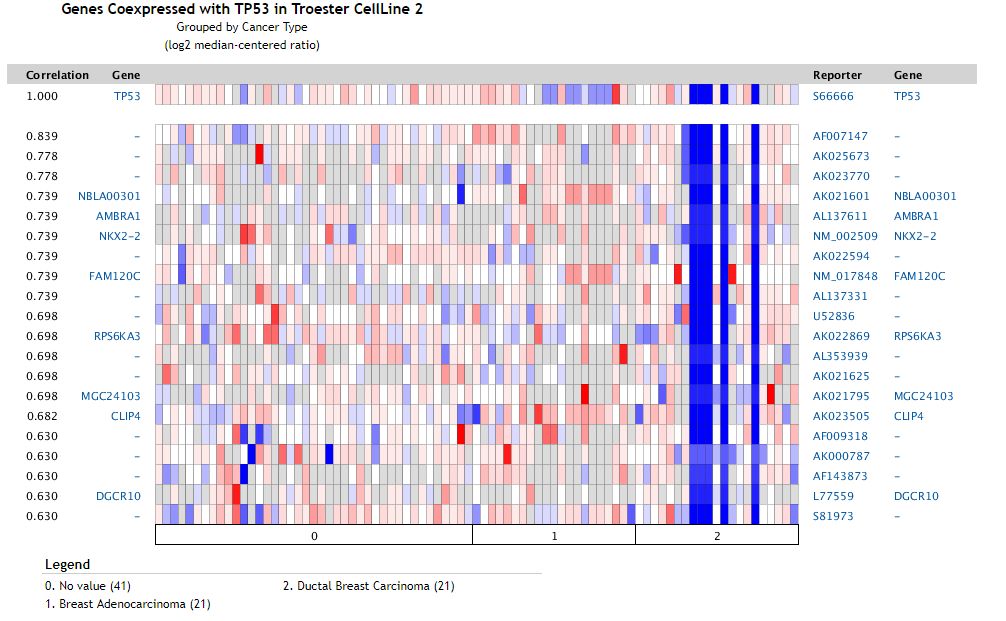
correlation从0到1,越接近于0, 说明相关性越高。
有以下两种差异分析
cancer_vs_cancer
cancer_vs_normal
差异分析的热图示意如下
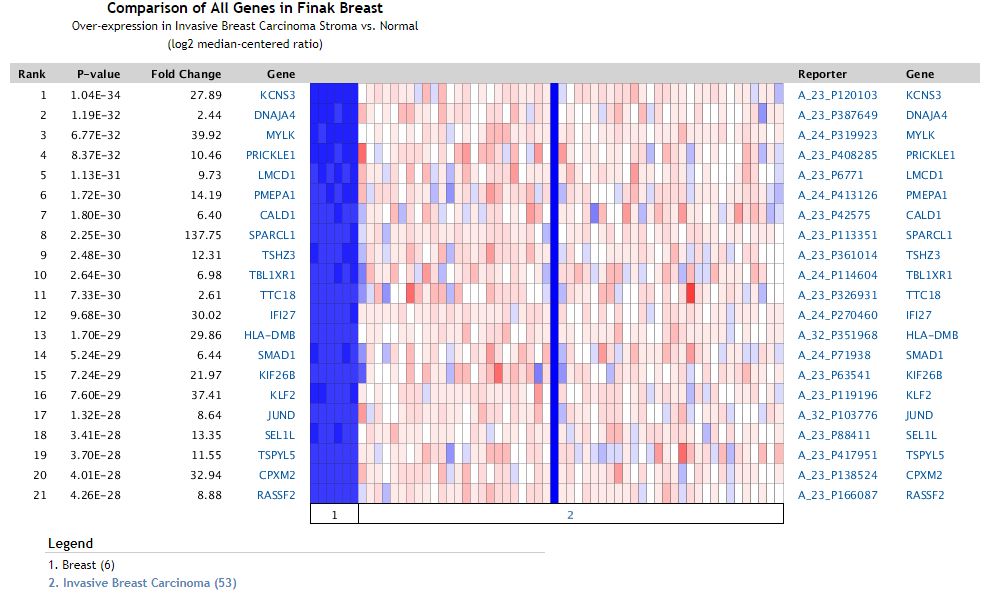
给出了差异的pvalue, fold change等统计学指标,对于多个基因的差异分析,用上述的热图形式展现,对于单个基因的差异分析结果,展现形式如下
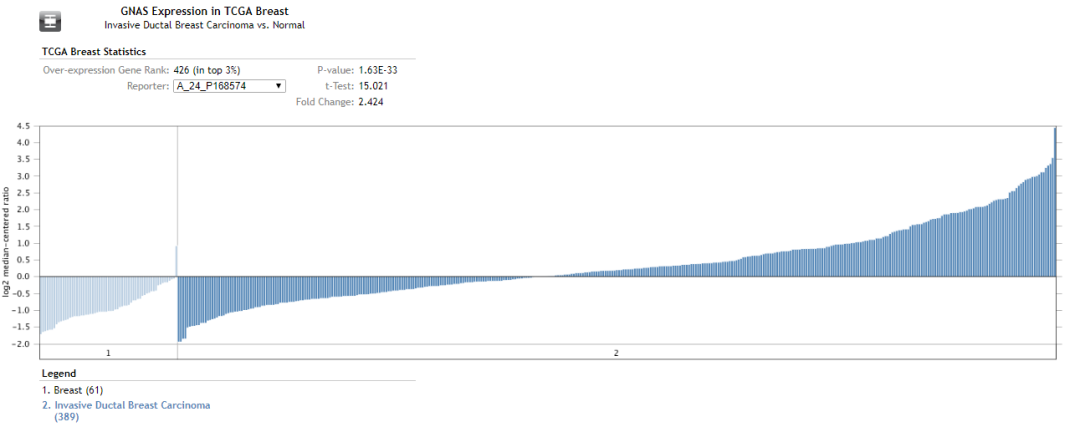
用柱状图的形式展示基因在每个样本中的表达量,每个柱子代表一个样本。
由于肿瘤样本的异质性,会出现原癌基因只在部分样本中过表达的例子,当用所有样本进行差异分析时,这部分基因的差异分析结果并不显著,为此专门开发了一种离群值分析的算法,全称如下
cancer outlier profile analysis
简称COPA, 来识别只在部分肿瘤样本中高表达的潜在的原癌基因。结果示意如下
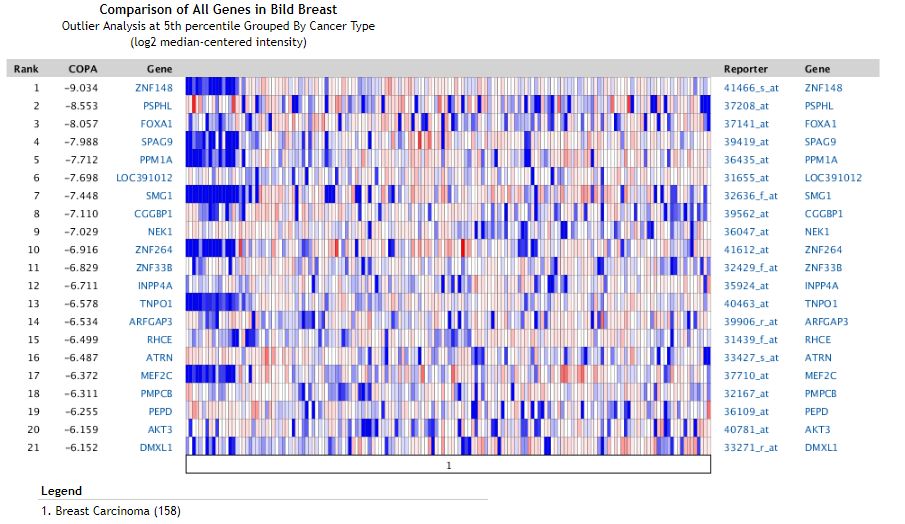
COPA值越高,说明该基因越可能是一个真实的离群基因。通过Oncomine数据库,可以方便的探究肿瘤相关的DNA拷贝数和基因表达谱数据。
以上是“Oncomine肿瘤芯片数据库的示例分析”这篇文章的所有内容,感谢各位的阅读!相信大家都有了一定的了解,希望分享的内容对大家有所帮助,如果还想学习更多知识,欢迎关注亿速云行业资讯频道!
亿速云「云数据库 MySQL」免部署即开即用,比自行安装部署数据库高出1倍以上的性能,双节点冗余防止单节点故障,数据自动定期备份随时恢复。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4580290/blog/4570460