怎么理解GWAS中的genotype imputation,针对这个问题,这篇文章详细介绍了相对应的分析和解答,希望可以帮助更多想解决这个问题的小伙伴找到更简单易行的方法。
GWAS用于寻找与疾病或者特定性状相关联的SNP位点,为了更加有效的挖掘信息,GWAS需要大样本量和高密度的SNP分型结果,最佳的分型方案当然是全基因组测序,然而成百上千个样本的全基因组测序其成本依然是巨大的,目前更加经济有效的方案是GWAS芯片,针对特定人群,利用tag SNP的思想设计探针,覆盖的SNP位点在几十M的数量级。
相比全基因组测序,GWAS芯片确实更加经济,但是其缺点也是显而易见的,只能够分析挖掘已知的SNP位点,而且位点数据量相对较少,要知道一个全基因组测序分析得到的SNP位点在几百M左右。为了解决这个问题,科学家提出了基因型填充的思想。
genotype imputation,称之为基因型填充,基本思想是利用单倍型来推断芯片未覆盖到的SNP位点的分型结果,在家系数据和独立样本的分析中都适用。家系样本基因型填充的过程示意如下

部分样本具有较为完整的SNP分型结果,依据这些样本的分型结果构建在家系样本中共享的单倍型,对应图中方框标记的完整分型结果,针对基因型缺失的样本,根据亲缘关系推断该样本可能的单倍型,对于基因型缺失的位点,直接使用对应单倍型中的分型结果进行填充。
独立样本的基因型填充过程示意如下
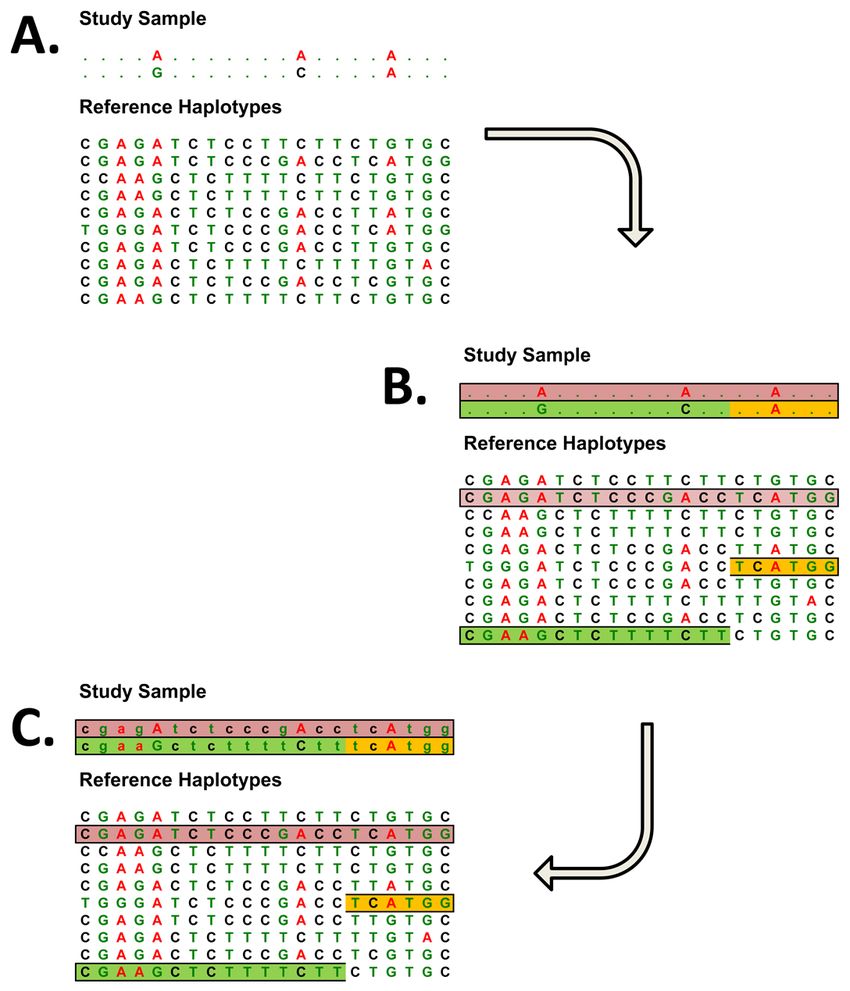
首先需要有一个参照的单倍型,根据样本已有的分型结果,与参照的单倍型进行比较,确定其可能所属的单倍型,然后进行填充。
以上示意图来自下列文献
Genotype Imputation
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC2925172/
从以上示意图可以看出,基因型填充有两个必要条件,第一个条件就是参照的单倍型,对于独立样本,可以采用hapmap或者1000G等项目的单倍型作为参照,第二个条件就是已知分型结果位点的比例和分布,对于需要填充的样本,要保证一定密度的分型结果,需要根据已有的分型结果来推断该样本可能的单倍型,分型结果已知的位点越多,其单倍型推断的准确性越高,填充的准确性才会越高,根据这个条件来看,GWAS芯片最适合进行基因型填充,因为其覆盖的SNP位点的数量和分布更有助于推测样本的单倍型。
目前已经有很多用于基因型填充的软件,部分列表如下
Beagle
IMPUTE2
MACH
关于怎么理解GWAS中的genotype imputation问题的解答就分享到这里了,希望以上内容可以对大家有一定的帮助,如果你还有很多疑惑没有解开,可以关注亿速云行业资讯频道了解更多相关知识。
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。