GtRNAdb:tRNA数据的示例分析,相信很多没有经验的人对此束手无策,为此本文总结了问题出现的原因和解决方法,通过这篇文章希望你能解决这个问题。
GtRNAdb收录了不同物种的tRNA序列信息,这些tRNA都是通过tRNAscan-SE 2.0软件预测得到,官网如下
http://gtrnadb.ucsc.edu/
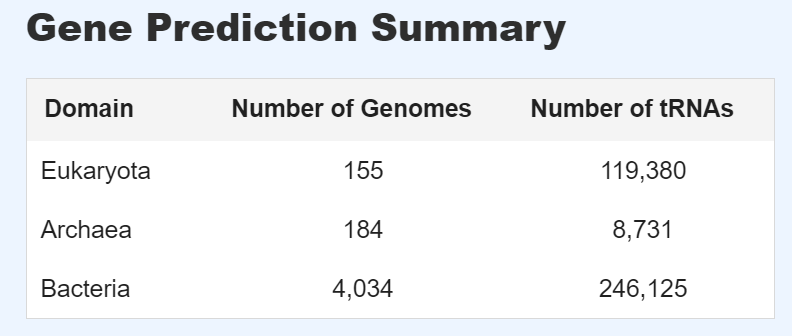
最新版本收录了超过4300个物种的tRNA序列,详细统计信息如下
通过该网站,我们可以检索物种对应的tRNA结果,也可以进行BLAST比对。
支持通过物种进行检索,根据进化关系,筛选感兴趣的物种,链接如下
http://gtrnadb.ucsc.edu/browse.html

点击三角符号展开,然后筛选相应的次级分类即可。以hg38为例,检索结果链接如下
http://gtrnadb.ucsc.edu/genomes/eukaryota/Hsapi38/
通过左侧的菜单栏,可以详细查看对应的信息。通过FASTA Seqs菜单,可以下载tRNA序列。

通过比对tRNA数据库,确定输入序列类型,链接如下
http://gtrnadb.ucsc.edu/blast.html
需要注意的是,在某些物种中,存在由tRNA衍生出来的重复序列,这些序列一级结构与tRNA非常接近,所以在tRNAscan-SE的预测结果中,会存在部分假阳性结果。GtRNAdb利用特定算法,对tRNAscan-SE直接预测的结果进行了一个过滤,降低了假阳性率。
对于数据库中已知的物种,建议直接利用数据库中的序列就好;对于数据库中没有的物种,再直接用tRNAscan-SE 进行预测。
看完上述内容,你们掌握GtRNAdb:tRNA数据的示例分析的方法了吗?如果还想学到更多技能或想了解更多相关内容,欢迎关注亿速云行业资讯频道,感谢各位的阅读!
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。