RepeatMasker中如何查找基因组上的重复序列,针对这个问题,这篇文章详细介绍了相对应的分析和解答,希望可以帮助更多想解决这个问题的小伙伴找到更简单易行的方法。
RepeatMasker软件用于查找基因组上的重复序列,默认情况下,会将重复序列原有的碱基用N代替,从而达到标记重复序列的目的。除此之外,也可以采用将重复序列转换为小写或者直接去除的方式,来标记重复序列。
该软件将输入的DNA序列与Dfam和Repbase数据库中已知的重复序列进行比对,从而识别输入序列中的重复序列。在比对时,支持以下多款软件
hmmer
cross_match
ABBlast/WUBlast
RMBlast
任选其中一个即可。官方提供了在线服务,网址如下
http://www.repeatmasker.org/cgi-bin/WEBRepeatMasker
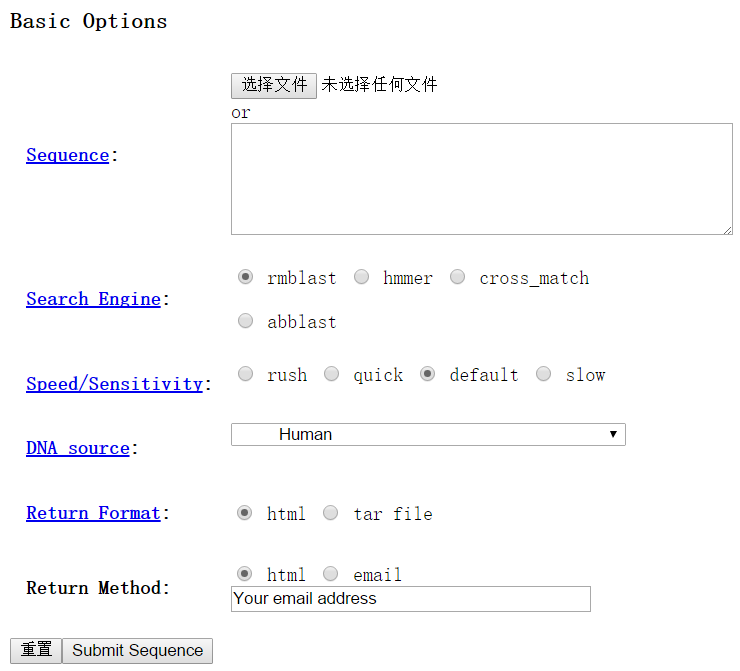
在Sequence中输入或者上传FASTA格式的DNA序列;Search Engine选择比对软件,Speed/Sensitivity选择运行模式,不同模式的主要区别在于运行速度与敏感度的差异,DNA source选择参考物种。
当然也可以下载软件到本地运行,安装过程如下
wget http://www.repeatmasker.org/RepeatMasker-open-4-0-7.tar.gz
tar xzvf RepeatMasker-open-4-0-7.tar.gz
cd RepeatMasker
perl ./configure需要注意的是,至少需要安装上述四种比对软件中的任意一种。此外,还需要安装TRF软件,链接如下
http://tandem.bu.edu/trf/trf.html
在安装过程中需要指定比对软件和TRF软件的安装位置。
软件基本用法如下
RepeatMasker -pa 5 -small -species human chrM.fa
-pa指定线程数,只有输入文件大于50Kb时才发挥作用;-small表示将重复序列转换为小写,-species指定参考的物种。
运行完成后,会生成多个文件,后缀为masked的文件为标记重复序列后的文件,后缀为.out的文件保存了重复序列区间信息。
关于RepeatMasker中如何查找基因组上的重复序列问题的解答就分享到这里了,希望以上内容可以对大家有一定的帮助,如果你还有很多疑惑没有解开,可以关注亿速云行业资讯频道了解更多相关知识。
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4580290/blog/4620836