小编给大家分享一下motif中PFM矩阵是怎么样的,希望大家阅读完这篇文章之后都有所收获,下面让我们一起去探讨吧!
PFM全称为position frequency matrix, 用于代表motif的碱基分布频数,本身是一个很容易了解的概念,以下图所示的motif序列为例
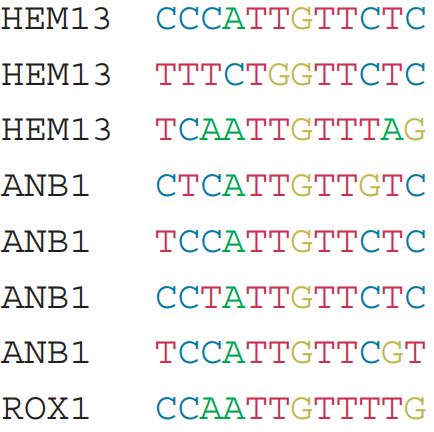
根据以上8条序列可以统计出对应的碱基分布频数,如下所示

每行为一种碱基,每一列为motif的一个位置。
在描述motif信息时,除了一致性序列和sequence logo外,PFM矩阵也是一个常见的元素。不同软件会有不同的标准,理解这些格式就是本文的核心内容。
JASPAR是一个常用的转录因子motif数据库,在该数据库中,针对PFM矩阵有多种格式,如下图所示
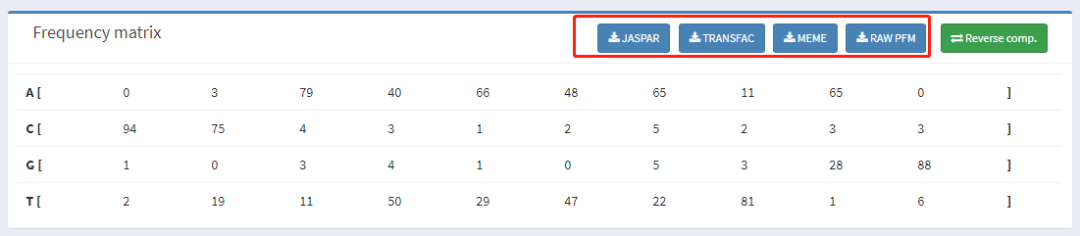
原始的PFM矩阵示意如下
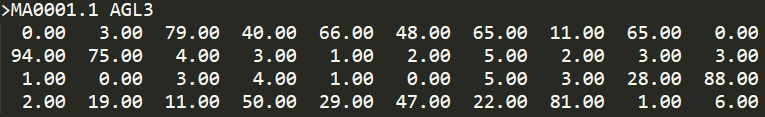
第一行和fasta格式的序列标识符类似,>开头,MA开头的字符串为转录因子在JASPAR数据库中的编号,是唯一的,AGL3表示该转录因子的名称。
接下来的4行依次表示A, C, G, T4种碱基在每个位置的频数分布。
JASPAR格式的PFM矩阵示意如下
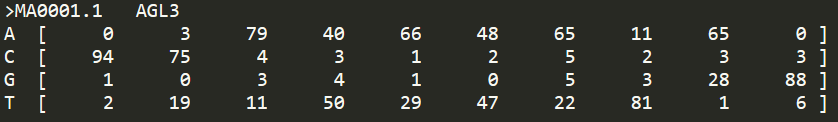
和原始的PFM矩阵非常类似,只不过在每行的开头标注了对应的碱基,并且用[和]操作符将碱基频数矩阵括起来。
TRANSFAC格式的PFM矩阵示意如下
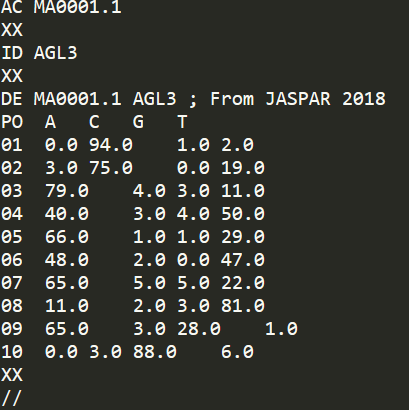
采用了TRANSFAC数据库中的文件标准,AC表示motif编号,ID表示motif的名称,PO以及下面的行为对应的碱基分布频数。
MEME格式的PFM矩阵示意如下
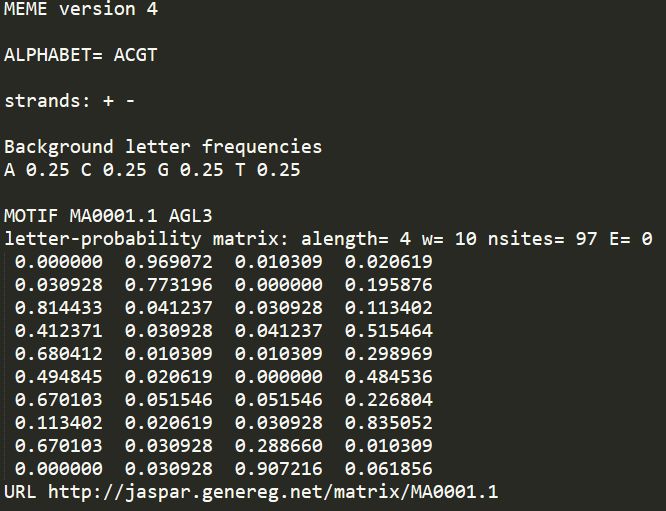
ALPJABEAT代表碱基的字符集,strands代表链的方向性,+ - 代笔在使用meme预测motif时没有指定链的方向,Background代表背景中的碱基组成频率,MOTIF和以下的行为对应的碱基分布频率。
不同的软件和数据库对应的PFM矩阵的格式不同,在使用不同软件和数据库时需要注意。
看完了这篇文章,相信你对“motif中PFM矩阵是怎么样的”有了一定的了解,如果想了解更多相关知识,欢迎关注亿速云行业资讯频道,感谢各位的阅读!
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。