这篇文章主要讲解了“如何使用seqLogo可视化motif”,文中的讲解内容简单清晰,易于学习与理解,下面请大家跟着小编的思路慢慢深入,一起来研究和学习“如何使用seqLogo可视化motif”吧!
seqLogo是一个bioconductor上的R包,专门用于DNA序列的motif可视化,网址如下
https://www.bioconductor.org/packages/release/bioc/html/seqLogo.html
因为功能的单一性,所以其用法也特别的简单,只需要输入motif对应的PPM矩阵就可以了,下面通过一个实际例子来看下

上图为一个motif的PFM矩阵,只需要通过以下几个步骤就可以得到对应的sequence logo。
将PFM矩阵保存在一个文件pfm.txt中,内容如下
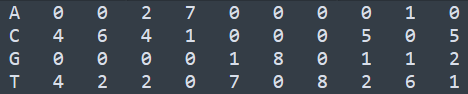
注意PFM矩阵中规定碱基顺序为ACGT, 不能任意调换碱基的顺序。通过R语言来读取,代码如下
data <- read.table("pfm.txt", header = F, sep = "\t", row.names = 1)PPM矩阵就是将PFM矩阵中的频数转化成频率,除以每列的总和就可以了, 代码如下
ppm <- sapply(1:ncol(data), function(t){ data[[t]] / sum(data[[t]]) })ppm的内容如下
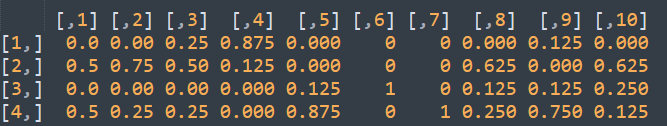
因为行的顺序统一为ACGT, 所以我们不需要设置行名称。
基本用法如下
p <- makePWM(ppm)
seqLogo(p)输出结果示意如下
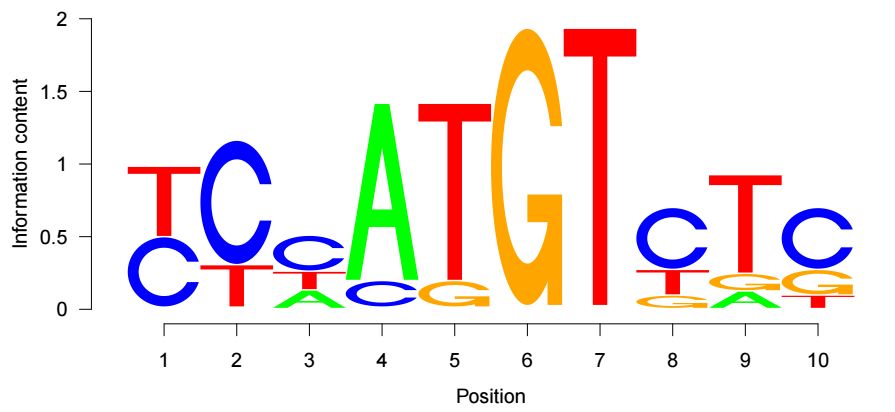
相比其他工具,这个R包的功能较为单一,存在无法调整配色方案等问题,但是作为一个可视化工具,其足够简单,已经能满足基本要求。
感谢各位的阅读,以上就是“如何使用seqLogo可视化motif”的内容了,经过本文的学习后,相信大家对如何使用seqLogo可视化motif这一问题有了更深刻的体会,具体使用情况还需要大家实践验证。这里是亿速云,小编将为大家推送更多相关知识点的文章,欢迎关注!
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。