这篇文章给大家介绍如何理解chromatin loops染色质,内容非常详细,感兴趣的小伙伴们可以参考借鉴,希望对大家能有所帮助。
利用5kb分辨率的Hi-C基因组互作图谱,科学家识别到了chromatin loop这种染色质结构,
通过对不同分辨率的Hi-C图谱进行分析,检测到了不同层级的染色质结构,从高到低依次为A/B compartments, subcompartments, TAD, chromation loop。Hi-C图谱和染色质结构模型的对应关系如下
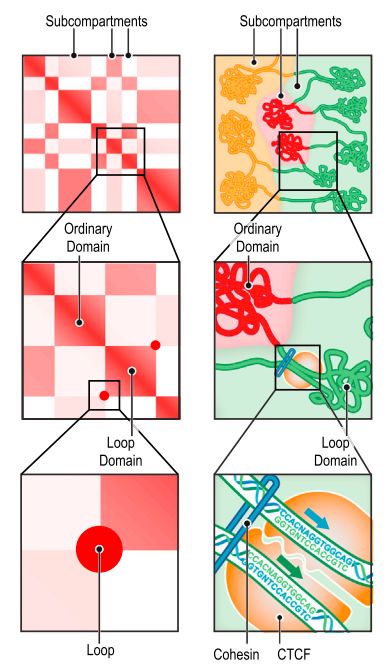
早期研究中利用1MB的Hi-C图谱 ,定义了每条染色质包含了A和B两个compartments。该文章中对100kb分辨率的HI-C图谱进行聚类分析,发现A/B compartments进一步分成了6个子类,即subcompartments。对每条染色质的Hi-C图谱进行不同算法的聚类分析,除了19号染色质外,都得到了5个cluster,对于19号染色质,得到了6个cluster。
通过分析这些subcompartments与A/B compartments的关系,发现其中2个属于A compartments, 标记为A1和A2, 另外4个属于B comprtmants, 标记为B1, B2, B3, B4。
对于TAD拓扑关联结构域的识别,首创了一种ArrowHead算法,核心是对归一化之后的交互矩阵进行变换,变化的公式如下
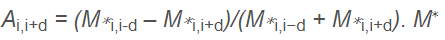
和DI的算法类似,i-d和i+d分别代表上下游的两个bin, 如果i和上游bin互作频率高,则A值为正数,如果和下游bin互作频率高,则A值为负数,如果相等,则为0。示意如下
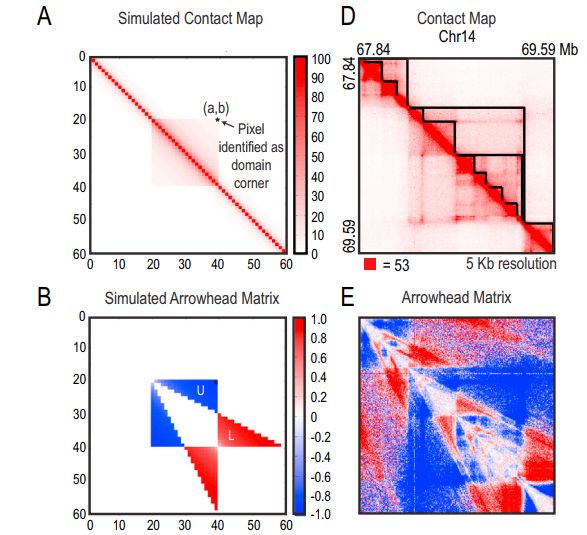
A图中的正方形区域为一个TAD domain, 经过转换后,变成了图B所示的形状。对应到整个互作矩阵上,看上去就是图E所示的箭头型。利用动态规划算法,识别变换后矩阵中的箭头区域,就可以预测TAD domain。文章中识别到的TAD domain,长度在40kb-3Mb,中位数为185kb。
对于染色质环,定义为Hi-C图谱中互作频率比周围相邻区域都高的格子区域,这样的区域称之为peak , 而对应的染色质区域称之为peak loci,如下图中蓝色圆点标记的区域
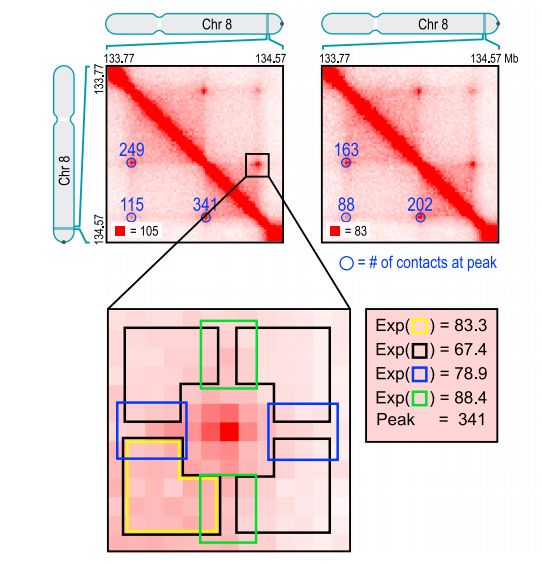
如上图所示,通过与四周区域的交互频率进行比较鉴定peak区域,这要去HI-C图谱的分辨率在5kb以下。对于全基因组互作图谱而言,这个计算量是非常大的,文章作者也提供了一种名为HiCCUPS的算法,集成在了开发的juicer软件中。
进一步比较不同细胞系和不同物种间的染色质环区域,结果如下
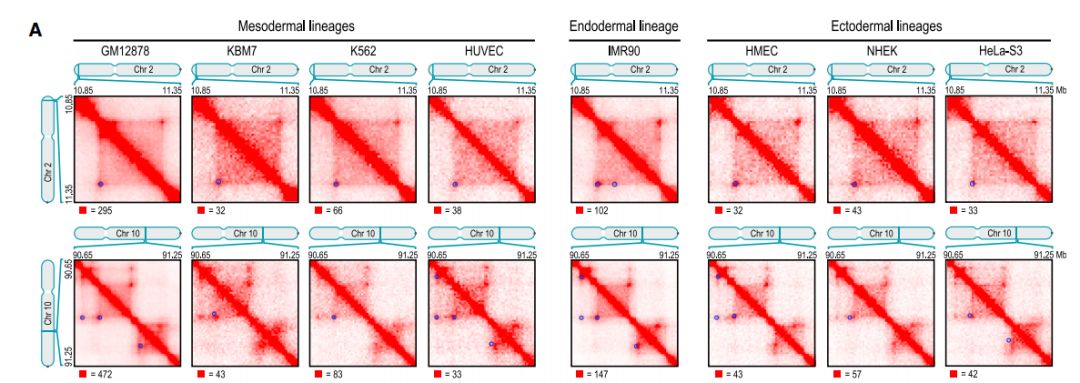
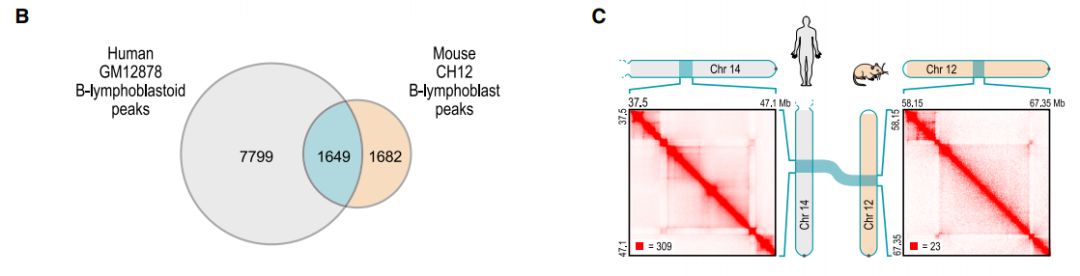
发现在不同细胞系间相对稳定,在物种间也具有一定进化保守性。对染色质环的分布位置进行分析,发现其处于TAD边界处。进一步分析发现染色质环中有很大部分为promoter-enhancer loops, 这也解释了增强子对靶基因的调控机制,虽然增强子与靶基因线性距离很远,但是增强子与靶基因启动子位于一个染色质环上,空间距离近,通过与启动子结合调控靶基因。
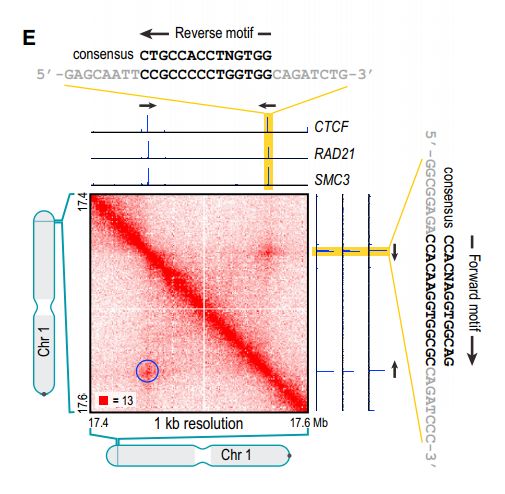
对染色质环对应区域富集的各种mark进行分析,发现其富集CTCF等转录因子, 如下图所示
对于染色质环的空间结构,提出了如下模型
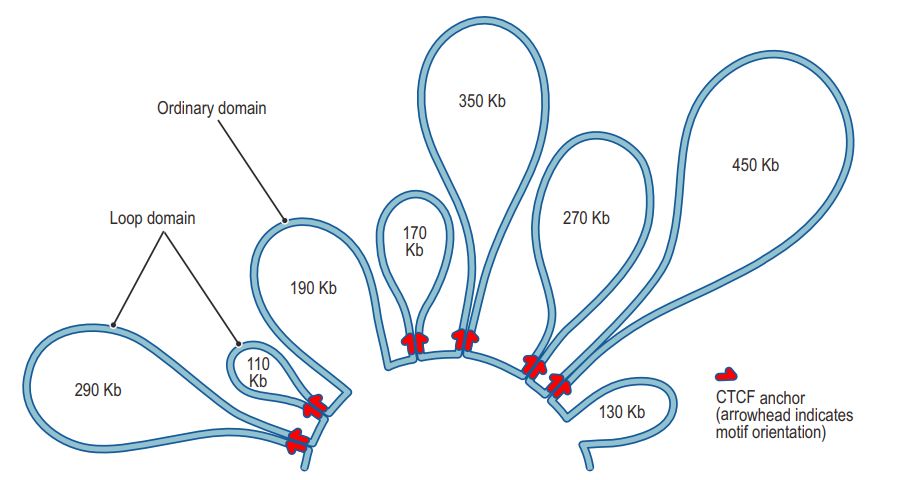
通过构建5kb以下分辨率的Hi-C图谱,可以识别染色质环这种染色质结构单元。
关于如何理解chromatin loops染色质就分享到这里了,希望以上内容可以对大家有一定的帮助,可以学到更多知识。如果觉得文章不错,可以把它分享出去让更多的人看到。
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。