R语言中的Anosim分析该如何理解,相信很多没有经验的人对此束手无策,为此本文总结了问题出现的原因和解决方法,通过这篇文章希望你能解决这个问题。
无论是野外环境样品,还是室内试验样品,一般我们都会设置样方或平行样来增强分析的准确性,必要时还会进行区组设计,因此在数据分析中需要进行组间差异的比较判别。然而对于微生物群落数据,由于物种繁多,而且不同物种的敏感环境因子不同,因此基于正态分布的参数检验难以满足分析需要,要进行多元非参数检验(non-parametric multivariate statistical tests)来计算显著性,R语言vegan包含有多种非参数检验方法,包括Anosim、Adonis、MRPP等,不同方法在统计量的选择、零模型等方面存在差异。
#读取抽平后的OTU_table和环境因子信息data=read.csv("otu_table.csv", header=TRUE, row.names=1)envir=read.table("environment.txt", header=TRUE)rownames(envir)=envir[,1]env=envir[,-1]#筛选高丰度物种并将物种数据标准化means=apply(data, 1, mean)otu=data[names(means[means>10]),]otu=t(otu)#根据地理距离聚类kms=kmeans(env, centers=3, nstart=22)Position=factor(kms$cluster)#进行Anosim分析library(vegan)anosim=anosim(otu, Position, permutations=999)summary(anosim)


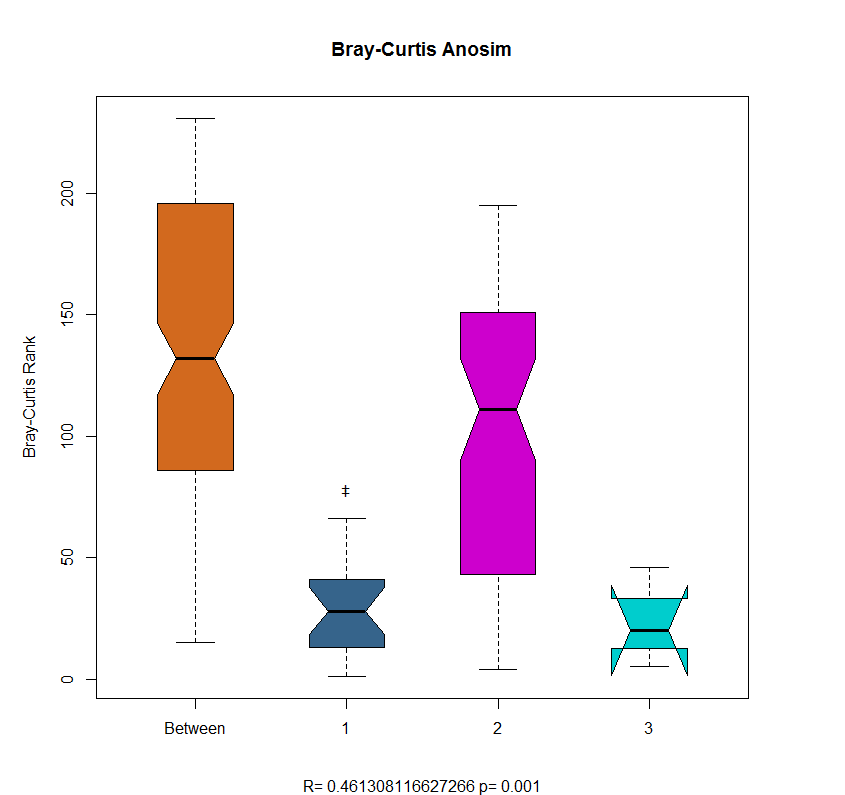
假如R>0,说明组内距离小于组间距离,也即分组是有效的,这与方差分析中比较组内方差与组间方差来判断的原理是类似的。由上面分析结果可以看到R=0.4613,大于零模型99%分位数0.290,因此p值为0.001,结果是显著的。我们可以提取分析结果,如下为距离的秩:

因为有22个样品,所以应该有C(22, 2)=231个距离。如下为上述距离对应的归属:

mycol=c(52,619,453,71,134,448,548,655,574,36,544,89,120,131,596,147,576)mycol=colors()[mycol]par(mar=c(5,5,5,5))result=paste("R=",anosim$statistic,"p=", anosim$signif)boxplot(anosim$dis.rank~anosim$class.vec, pch="+", col=mycol, range=1, boxwex=0.5, notch=TRUE, ylab="Bray-Curtis Rank", main="Bray-Curtis Anosim", sub=result)
看完上述内容,你们掌握R语言中的Anosim分析该如何理解的方法了吗?如果还想学到更多技能或想了解更多相关内容,欢迎关注亿速云行业资讯频道,感谢各位的阅读!
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。