这篇文章主要讲解了“如何使用IMPUTE2进行基因型填充”,文中的讲解内容简单清晰,易于学习与理解,下面请大家跟着小编的思路慢慢深入,一起来研究和学习“如何使用IMPUTE2进行基因型填充”吧!
提供了以下两大功能
haplotype phasing,单倍型分析
genotype imputation,基因型填充
基因型填充的基本模型示意如下
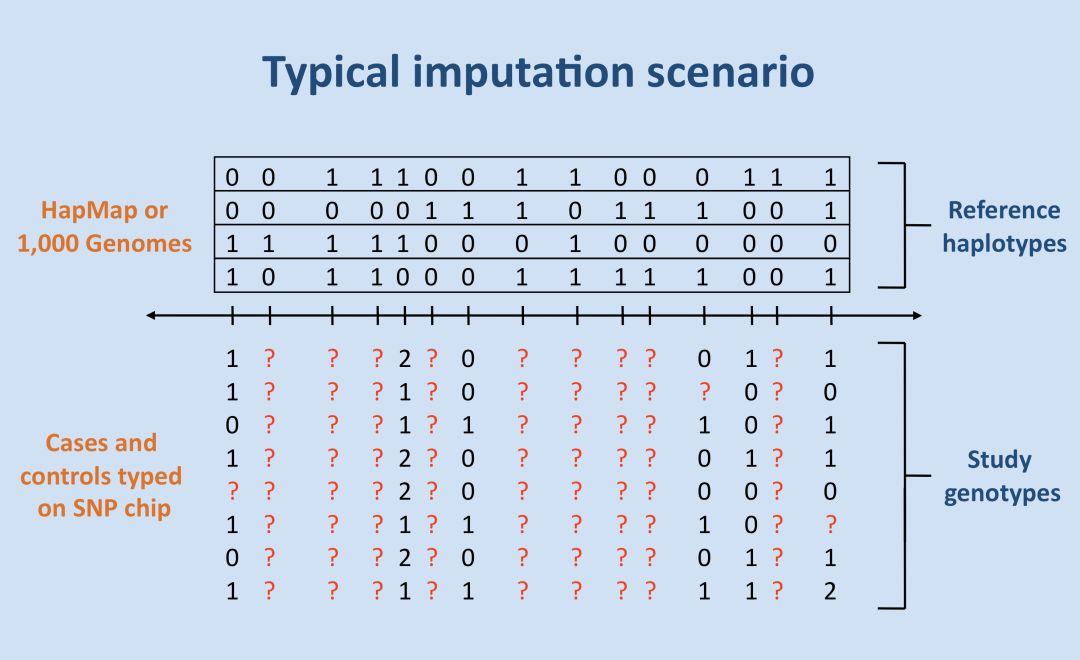
需要两个基本元素,第一个是检测样本的分型结果,即图中所示的study genotypes, 第二个元素称之为reference panel, 对应图中的reference haplotypes, 利用高密度的reference panel对检验样本为覆盖到的SNP位点,或者缺失的分型结果进行填充,对应图中问号表示的位点。
该软件的安装比较简单,官网提供了编译好的可执行文件,下载解压缩即可
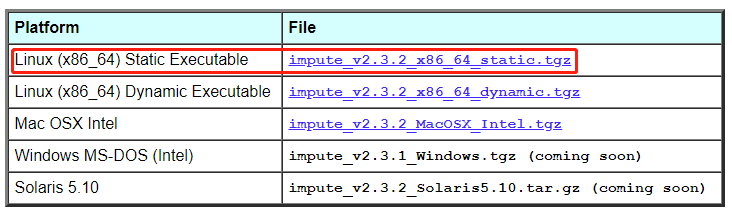
对应的代码如下
wget https://mathgen.stats.ox.ac.uk/impute/impute_v2.3.2_x86_64_static.tgztar xzvf impute_v2.3.2_x86_64_static.tgz
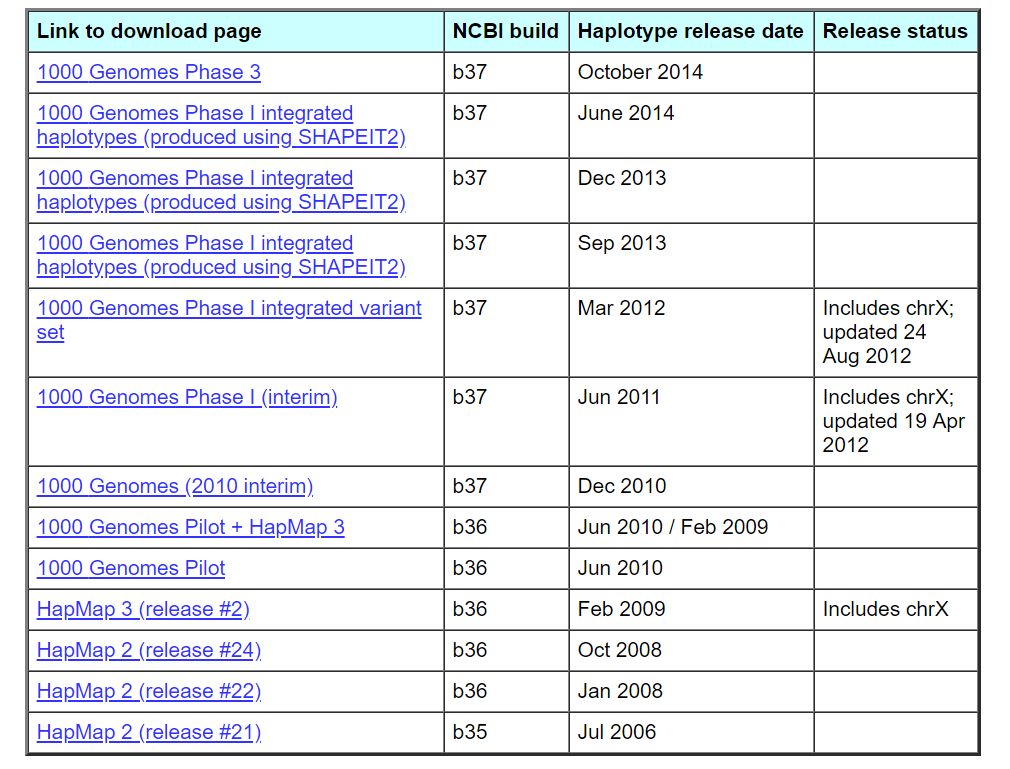
除了软件外,还需要reference panel,官网也提供了对应的下载文件,包括hapmap和1000G两个常用的reference panel,链接如下
https://mathgen.stats.ox.ac.uk/impute/impute_v2.html#download
impute2官方推荐了一套基因型填充的最佳实践,步骤如下
对检测样本的原始分型结果质控,使用GWAS分析的质控条件即可
校正基因组版本,hapmap和1000G都是基于hg19版本,必须保证和reference panel的基因组版本一致,才可以准确填充,如果不一致,可以使用UCSC的liftOver工具进行转换
校正链的方向, hapmap和1000G的结果都是基于参考基因组的正链表示的,为了和reference panel进行匹配,必须将芯片的分型结果也统计校正到正链
选择reference panel,1000G比hapmap的snp位点数量更多,密度更大,是目前最常用的reference panel, 其中又分成了不同的人群,对于某些研究,可以选择更加契合自己的人群,比如选择亚洲人群进行分析
基因型填充
填充后的质控,对填充后的分型结果进行过滤,同样基于GWAS的质控条件
关联分析,填充后的snp位点数量更多,有助于检测阳性的信号
显著关联区域的重新填充,对于GWAS筛选出来的阳性区域,可以使用更加严格的参数重新填充,再进行关联分析,确保分析的可靠性
impute2提供了以下两种用法
填充准确率最高的方法,基本用法如下
impute2 \-m ./Example/example.chr22.map \-h ./Example/example.chr22.1kG.haps \-l ./Example/example.chr22.1kG.legend \-g ./Example/example.chr22.study.gens \-strand_g ./Example/example.chr22.study.strand \-int 20.4e6 20.5e6 \-Ne 20000 \-o ./Example/example.chr22.one.phased.impute2
基因型填充计算量非常大,所以需要先拆分染色体,对每条染色体进行填充。上述是官方自带的一个例子,对22号染色体进行填充,-m参数指定连锁图谱,-h和-l参数指定reference panel的单倍型结果,对应后缀为haps和legend,-g参数指定study样本的分型结果,格式为GEN, -strand_g参数指定snp位点的正负链信息,用于校正链的方向,-int参数指定需要填充的染色体区域,包含了起始和终止两个位置的值,对应的长须推荐小于5Mb, -Ne参数官方推荐取值为20000,-o参数指定输出的填充结果。
增加了对study样本的pre-phasing, 运行速度更快,pre-phasing基本用法如下
impute2 \-prephase_g \-m ./Example/example.chr22.map \-g ./Example/example.chr22.study.gens \-int 20.4e6 20.5e6 \-Ne 20000 \-o ./Example/example.chr22.prephasing.impute2
-prephase_g参数表示对study样本进行pre-phasing, -m参数可以提高单倍型分析的准确性。
基于pre-phasing的结果进行填充的基本用法如下
./impute2 \-use_prephased_g \-m ./Example/example.chr22.map \-h ./Example/example.chr22.1kG.haps \-l ./Example/example.chr22.1kG.legend \-known_haps_g ./Example/example.chr22.prephasing.impute2_haps \-strand_g ./Example/example.chr22.study.strand \-int 20.4e6 20.5e6 \-Ne 20000 \-o ./Example/example.chr22.one.phased.impute2-phase
即使采用两步法,基因型填充仍然是一个运行时间很长的步骤,在实际操作中,可以同时结合染色体拆分和染色体划分窗口两种方式,再加上并行来提升运行效率。
官方提供了更多的用法示例,链接如下
https://mathgen.stats.ox.ac.uk/impute/impute_v2.html#examples
感谢各位的阅读,以上就是“如何使用IMPUTE2进行基因型填充”的内容了,经过本文的学习后,相信大家对如何使用IMPUTE2进行基因型填充这一问题有了更深刻的体会,具体使用情况还需要大家实践验证。这里是亿速云,小编将为大家推送更多相关知识点的文章,欢迎关注!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4580290/blog/4357163