这篇文章主要介绍“如何使用R包SomaticSignatures进行denovo的signature推断”,在日常操作中,相信很多人在如何使用R包SomaticSignatures进行denovo的signature推断问题上存在疑惑,小编查阅了各式资料,整理出简单好用的操作方法,希望对大家解答”如何使用R包SomaticSignatures进行denovo的signature推断”的疑惑有所帮助!接下来,请跟着小编一起来学习吧!
原文在:http://bioconductor.org/packages/release/bioc/vignettes/SomaticSignatures/inst/doc/SomaticSignatures-vignette.html
library(SomaticSignatures)library(SomaticCancerAlterations)library(BSgenome.Hsapiens.1000genomes.hs37d5)sca_metadata = scaMetadata()sca_metadatasca_data = unlist(scaLoadDatasets())sca_data$study = factor(gsub("(.*)_(.*)", "\\1", toupper(names(sca_data))))sca_data = unname(subset(sca_data, Variant_Type %in% "SNP"))sca_data = keepSeqlevels(sca_data, hsAutosomes(), pruning.mode = "coarse")sca_vr = VRanges( seqnames = seqnames(sca_data), ranges = ranges(sca_data), ref = sca_data$Reference_Allele, alt = sca_data$Tumor_Seq_Allele2, sampleNames = sca_data$Patient_ID, seqinfo = seqinfo(sca_data), study = sca_data$study)sca_vr可以看到,这个包,需要的是sca_data这个变量里面各个列,用到了的就是 c( "Sample","chr", "pos","ref", "alt") 这些列。所以我们自己的somatic突变信息,也需要制作成为这5列。
在文章主页下载;https://static-content.springer.com/esm/art%3A10.1038%2Fs41422-020-0333-6/MediaObjects/41422_2020_333_MOESM23_ESM.csv
这个是大于500M的CSV文件,下载后修改名字,然后读入R,并且制作成为 SomaticSignatures 包的输入数据的代码如下:
library(data.table)b=fread('../maf.csv',data.table = F)b[1:4,1:3]colnames(b)mut=btable(mut$Variant_Type)mut=mut[mut$Variant_Type=='SNP',]a=mut[,c(10,2,3,8,9)]colnames(a)=c( "Sample","chr", "pos","ref", "alt") alls=as.character(unique( a$Sample)) a$study=a$Samplehead(a)虽然我们使用了 data.table 包的 fread函数,可以超级快的读入大于500M的CSV文件,但是也需要一点时间啦。
制作的a这个变量,如下:
> head(a) Sample chr pos ref alt study2 FP1705100059DN01 chr1 4870770 G T FP1705100059DN013 FP1705100059DN01 chr1 5111686 C T FP1705100059DN014 FP1705100059DN01 chr1 5116099 C T FP1705100059DN015 FP1705100059DN01 chr1 5151401 C T FP1705100059DN016 FP1705100059DN01 chr1 5151403 G C FP1705100059DN017 FP1705100059DN01 chr1 5217189 G A FP1705100059DN01一个很普通的数据框而已,并不是SomaticSignatures 包的文档介绍sca_data这个变量的类型,但是该有的5列信息是有的。
sca_vr = VRanges( seqnames = a$chr , ranges = IRanges(start = a$pos,end = a$pos+1), ref = a$ref, alt = a$alt, sampleNames = as.character(a$Sample), study=as.character(a$study))sca_vr在SomaticSignatures 包已经是封装好的函数,很容易就可以获取,而且速度超级快哦,代码如下:
# 突变位点坐标基于hg19,从基因组根据坐标获取碱基上下文sca_motifs = mutationContext(sca_vr, BSgenome.Hsapiens.UCSC.hg19)head(sca_motifs)# 对每个样本,计算 96 突变可能性的 比例分布情况escc_sca_mm = motifMatrix(sca_motifs, group = "study", normalize = TRUE)dim( escc_sca_mm )table(colSums(escc_sca_mm))head(escc_sca_mm[,1:4])我们都知道,sanger研究所科学家【1】提出来了肿瘤somatic突变的signature概念 ,把96突变频谱的非负矩阵分解后的30个特征,在cosmic数据库可以学习它。不同的特征有不同的生物学含义【2】,比如文章【3】 就是使用了 这些signature区分生存!主要是R包deconstructSigs可以把自己的96突变频谱对应到cosmic数据库的30个突变特征。
但是我们现在要自己推断denovo的signature,所以使用SomaticSignatures 包的identifySignatures函数哦,代码如下:
# 预先设定待探索的 signature 数量范围,文章最后选定11个if(F){ n_sigs = 5:15 gof_nmf = assessNumberSignatures(escc_sca_mm , n_sigs, nReplicates = 5) save(gof_nmf,file = 'gof_nmf.Rdata')}load(file = 'gof_nmf.Rdata')# 这个 assessNumberSignatures 步骤耗时很严重。plotNumberSignatures(gof_nmf)# 根据这个图表,选择11个 signature sigs_nmf = identifySignatures(escc_sca_mm , 11, nmfDecomposition)save(escc_sca_mm,sigs_nmf,file = 'escc_denovo_results.Rata')代码如下:
load(file = 'escc_denovo_results.Rata')str(sigs_nmf) library(ggplot2)plotSignatureMap(sigs_nmf) + ggtitle("Somatic Signatures: NMF - Heatmap")plotSignatures(sigs_nmf, normalize =T) + ggtitle("Somatic Signatures: NMF - Barchart") + facet_grid(signature ~ alteration,scales = "free_y") 出图如下:
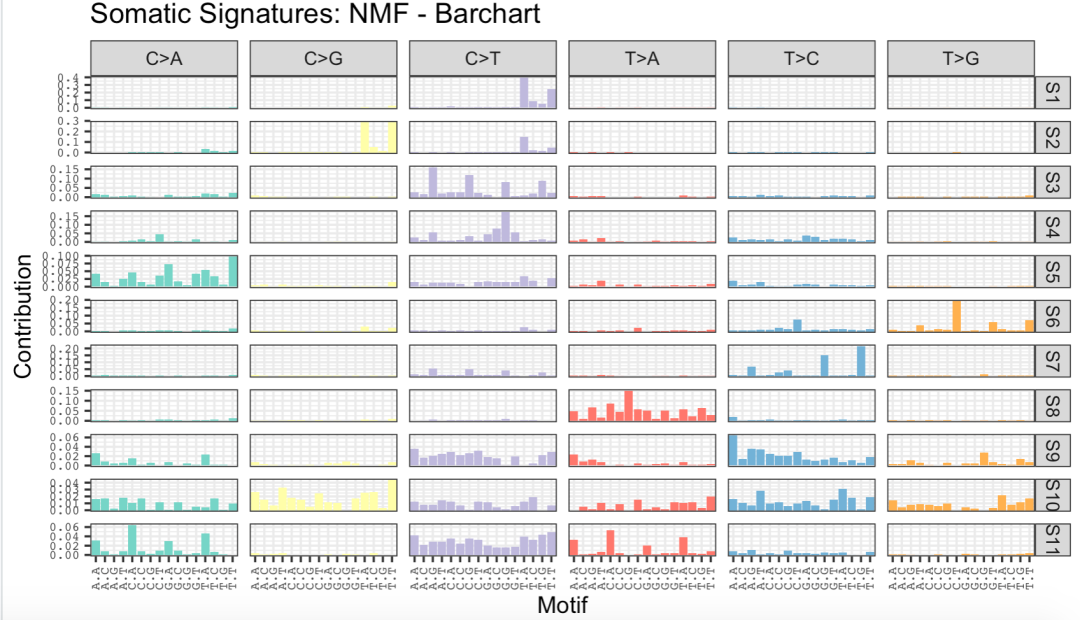
到此,关于“如何使用R包SomaticSignatures进行denovo的signature推断”的学习就结束了,希望能够解决大家的疑惑。理论与实践的搭配能更好的帮助大家学习,快去试试吧!若想继续学习更多相关知识,请继续关注亿速云网站,小编会继续努力为大家带来更多实用的文章!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4503882/blog/4352319