这篇文章主要介绍R语言如何实现可视化展示gff3格式基因组注释文件,文中介绍的非常详细,具有一定的参考价值,感兴趣的小伙伴们一定要看完!
使用R语言的 ggbio 这个包可视化gff3格式的基因组注释文件
我用到的文件是NCBI下载的拟南芥注释文件,为了减小计算压力,我只用到了gff文件的前119行,两个基因。
用到的函数是 GenomicFeatures R包中的 **makeTxDbFromGFF()**函数
library(GenomicFeatures)txdb<-makeTxDbFromGFF(file="practice.gff",format="gff3")用到的 ggbio 这个包中的 **autoplot()**这个函数
library(ggbio)autoplot(txdb, which=GRanges("CP002684.1", IRanges(100, 9000)), names.expr = "gene_id")+ theme_bw()结果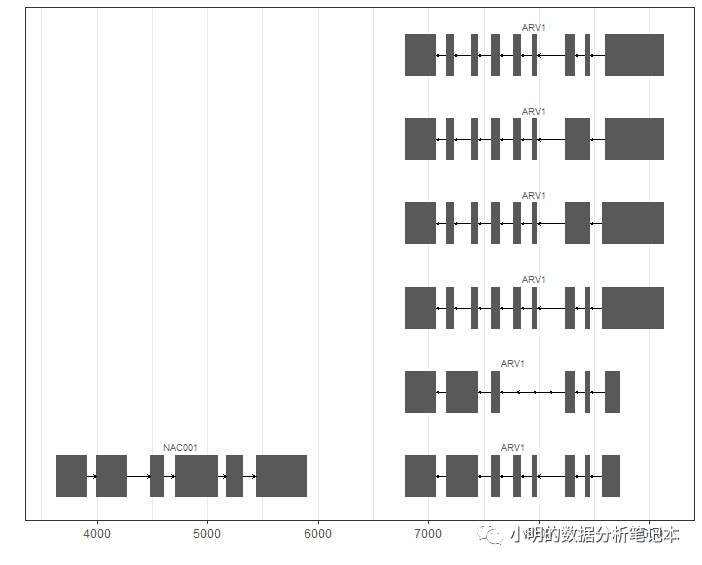
可以通过fill参数设置不同的颜色
autoplot(txdb, which=GRanges("CP002684.1", IRanges(100, 9000)), names.expr = "gene_id",fill="red")+ theme_bw()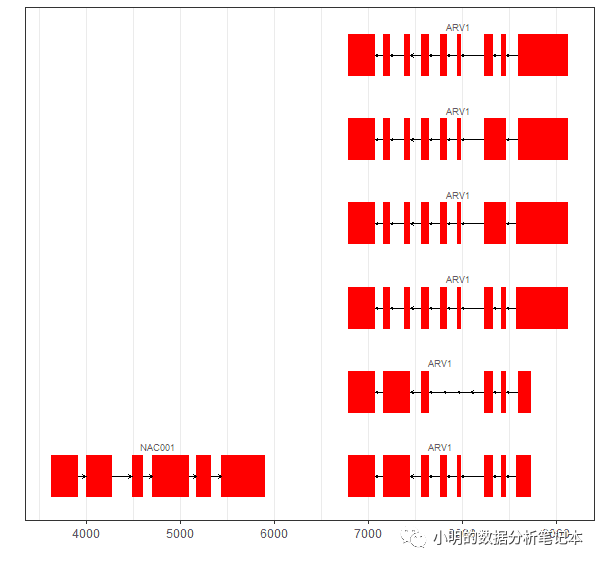
不同的基因填充不同的颜色
autoplot(txdb, which=GRanges("CP002684.1", IRanges(100, 9000)), names.expr = "gene_id",aes(fill=gene_id))+ theme_bw()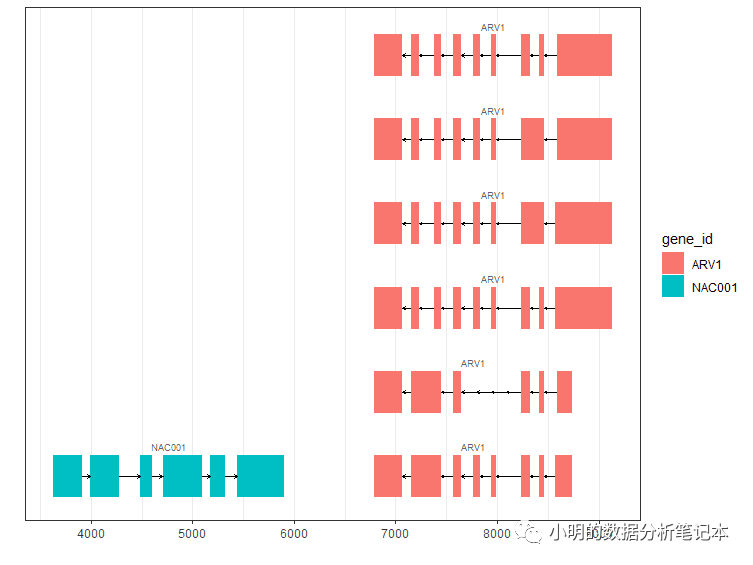
现在还不知道如何给同一个基因不同的部分(utr,exon,intron)等填充不同的颜色 还有就是 makeTxDbFromGFF() 函数读入的数据存储格式还没搞懂
开头提到的参考资料里有一幅图将 reads数量, 覆盖度的折线图,vcf文件的结果,gff可视化的结果画到了一起,做基因组重测序分析应该会用得到。这里暂时不重复了。等用到的时候再说。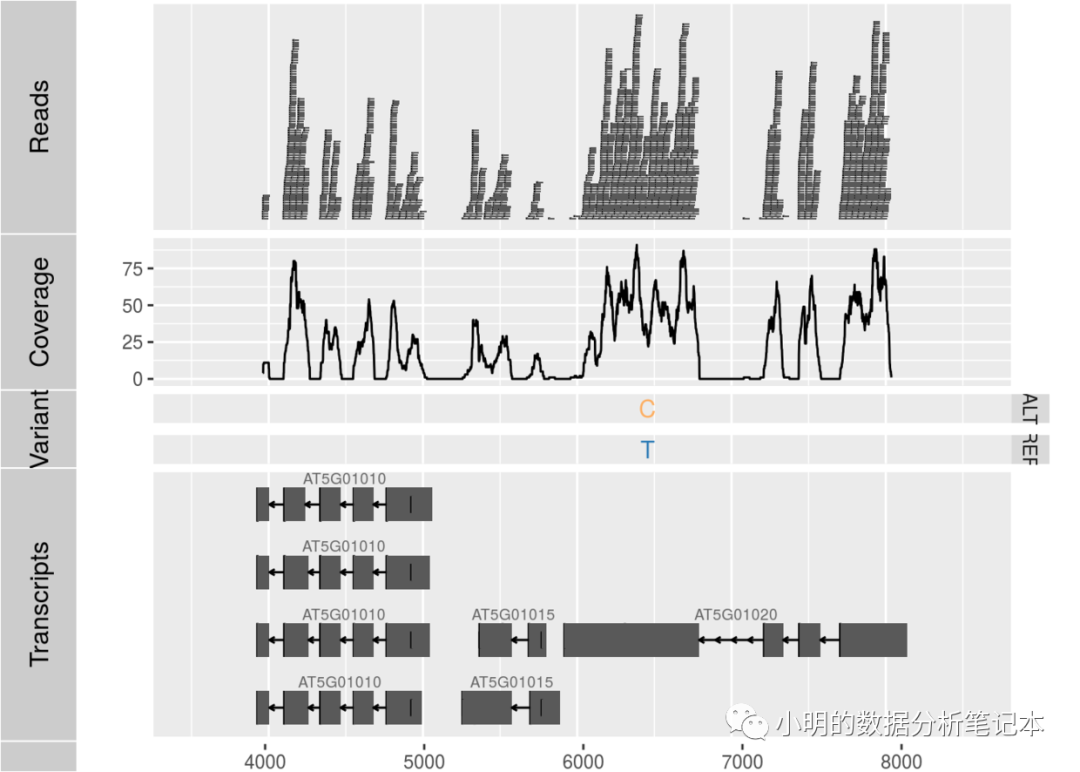
以上是“R语言如何实现可视化展示gff3格式基因组注释文件”这篇文章的所有内容,感谢各位的阅读!希望分享的内容对大家有帮助,更多相关知识,欢迎关注亿速云行业资讯频道!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4579431/blog/4347377