这篇文章将为大家详细讲解有关java如何实现基因序列比较,小编觉得挺实用的,因此分享给大家做个参考,希望大家阅读完这篇文章后可以有所收获。
设计算法,计算两给定基因序列的相似程度。
人类基因由4种核苷酸,分别用字母ACTG表示。要求编写一个程序,按以下规则比较两个基因序列并确定它们的相似程度。即给出两个基因序列AGTGATG和GTTAG,它们有多相似呢?测量两个基因相似度的一种方法称为对齐。使用对齐方法可以在基因的适当位置加入空格,让两个基因的长度相等,然后根据基因的分值矩阵计算分数。
看了很多代码基本上都是用c++或者c写的,但是习惯性写java就用java实现一下
基本的思路就是,和背包问题差不多,实现还是模仿填表的形式去实现的
表达式:
s1 = result[i-1][j-1] + getScore(X[i], Y[j]) 这个是x,y序列使用坐标匹配
s2 = result[i-1][j] + getScore(X[i], ‘-') 这个是x序列匹配y的 ‘-'
s3 = result[i][j-1] + getScore('-', Y[j]) 这个是y序列匹配x的 ‘-'
result[i][j] = max(s1,s2,s3) 找出三个中最大的就是所求的值
package algorithmClassSet.three;
import java.util.HashMap;
import java.util.Map;
/**
* s1 = result[i-1][j-1] + getScore(X[i], Y[j]) 这个是x,y序列使用坐标匹配
* s2 = result[i-1][j] + getScore(X[i], '-') 这个是x序列匹配y的 ‘-'
* s3 = result[i][j-1] + getScore('-', Y[j]) 这个是y序列匹配x的 ‘-'
* result[i][j] = max(s1,s2,s3) 找出三个中最大的就是所求的值
* m*n
*/
public class GeneSequenceComparison {
public static void main(String[] args) {
dealIt();
}
private static void dealIt() {
String[] X = {"A", "G", "T", "G", "A", "T", "G"};
String[] Y = {"G", "T", "T", "A", "G"};
int m = X.length + 1;
int n = Y.length + 1;
int[][] result = new int[m][n];
for (int i = 1; i < m; i++) {
result[i][0] = result[i - 1][0] + getScore(X[i - 1], "-");
}
for (int j = 1; j < n; j++) {
result[0][j] = result[0][j - 1] + getScore("-", Y[j - 1]);
}
for (int i = 1; i < m; i++) {
for (int j = 1; j < n; j++) {
int s1 = result[i - 1][j - 1] + getScore(X[i - 1], Y[j - 1]);
int s2 = result[i - 1][j] + getScore(X[i - 1], "-");
int s3 = result[i][j - 1] + getScore("-", Y[j - 1]);
int maxs = getMax(s1, s2, s3);
result[i][j] = maxs;
}
}
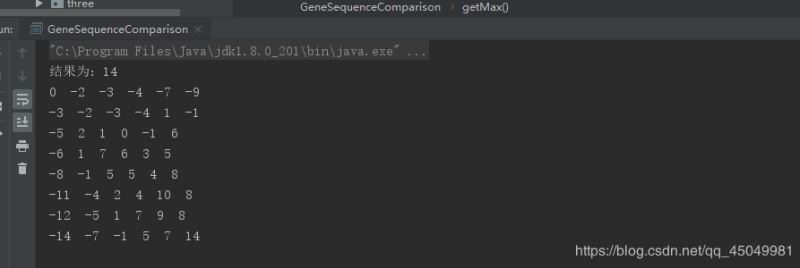
System.out.println("结果为:" + result[m - 1][n - 1]);
for (int i = 0; i < m; i++) {
for (int j = 0; j < n; j++) {
System.out.print(result[i][j] + " ");
}
System.out.println();
}
}
private static int getMax(int s1, int s2, int s3) {
int flag = s1;
if (flag < s2) {
flag = s2;
}
if (flag < s3) {
flag = s3;
}
return flag;
}
//传入值获取分数
private static int getScore(String x, String y) {
//x和y必须属于 ACGT-
Map<String, Integer> map = new HashMap<>();
map.put("A", 0);
map.put("C", 1);
map.put("G", 2);
map.put("T", 3);
map.put("-", 4);
int[][] score = {
{5, -1, -2, -1, -3},
{-1, 5, -3, -2, -4},
{-2, -3, 5, -2, -2},
{-1, -2, -2, 5, -1},
{-3, -4, -2, -1, -10000000}};
return score[map.get(x)][map.get(y)];
}
}
关于“java如何实现基因序列比较”这篇文章就分享到这里了,希望以上内容可以对大家有一定的帮助,使各位可以学到更多知识,如果觉得文章不错,请把它分享出去让更多的人看到。
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。