这篇文章将为大家详细讲解有关R语言如何实现柱状图排序和x轴上的标签倾斜操作,小编觉得挺实用的,因此分享给大家做个参考,希望大家阅读完这篇文章后可以有所收获。
R语言做柱状图大致有两种方法, 一种是基础库里面的 barplot函数, 另一个就是ggplot2包里面的geom_bar
此处用的是字符变量 统计其各频数,然后做出其柱状图。(横轴上的标签显示不全)
t <- sort(table(dat1$L), decreasing = TRUE) #将频数表进行排序
r <- barplot(t, col = "blue",
main = "柱状图", ylim = c(0,12), names.arg = dimnames(t) #画字符变量的柱状图
tmp <- as.vector(t) #将频数变成一个向量
text(r, tmp, label = tmp, pos = 3) #加柱子上面的标签或用ggplot2包 (目前仍没有给柱子上加数字标签)
library(ggplot2) #加载ggplot2包
reorder_size <- function(x) {
factor(x, levels = names(sort(table(x))))
} #自定义函数,获取因子型变量的因子类型
p <- ggplot(dat3, aes(reorder_size(LAI))) + #用因子变量做基础底图,也可直接用reorder排序
geom_bar(fill = "blue") + #画柱状图
theme(axis.text.x = element_text(angle = 45, hjust = 0.5, vjust = 0.5)) + #让横轴上的标签倾斜45度
xlab("柱状图") #给x轴加标签补充:R 语言条形图,解决x轴文字排序问题
数据结果的图形展示,R代码,《R数据科学》是个好东西
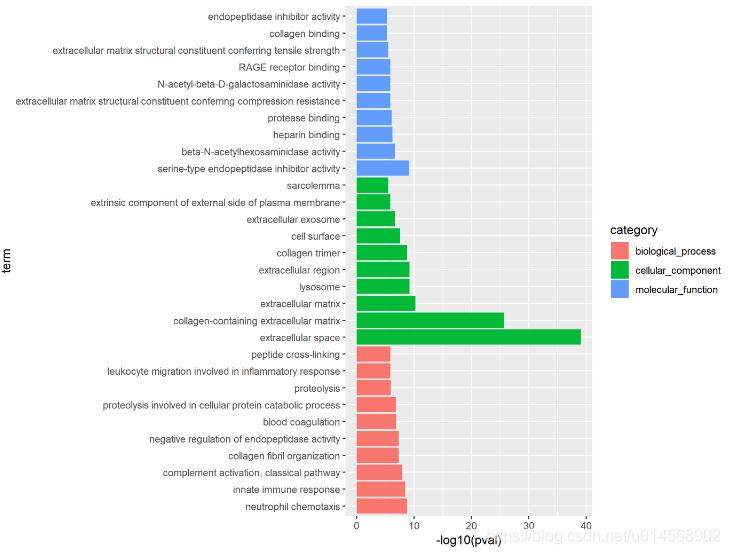
| term | category | pval |
| neutrophil chemotaxis | biological_process | 1.68E-09 |
| innate immune response | biological_process | 3.35E-09 |
| complement activation, classical pathway | biological_process | 1.14E-08 |
| negative regulation of endopeptidase activity | biological_process | 4.43E-08 |
| collagen fibril organization | biological_process | 4.43E-08 |
| blood coagulation | biological_process | 1.29E-07 |
| proteolysis involved in cellular protein catabolic process | biological_process | 1.56E-07 |
| proteolysis | biological_process | 1.13E-06 |
| leukocyte migration involved in inflammatory response | biological_process | 1.47E-06 |
| peptide cross-linking | biological_process | 1.47E-06 |
| extracellular space | cellular_component | 8.75E-40 |
| collagen-containing extracellular matrix | cellular_component | 2.08E-26 |
| extracellular matrix | cellular_component | 5.72E-11 |
| lysosome | cellular_component | 6.09E-10 |
| extracellular region | cellular_component | 6.58E-10 |
| collagen trimer | cellular_component | 1.68E-09 |
| cell surface | cellular_component | 2.80E-08 |
| extracellular exosome | cellular_component | 2.34E-07 |
| extrinsic component of external side of plasma membrane | cellular_component | 1.47E-06 |
| sarcolemma | cellular_component | 3.16E-06 |
作图要求:x轴为term,颜色按categroy分类、并且pval由小到大排序
#openxlsx读入为data.frame
class(data)
#转换
library(tidyverse)
godata<-as_tibble(godata)
class(godata)
#原始数据筛选(category,term,pval)散列,按照category,-log10(pval)排序
data<-godata%>%select(category,term,pval)%>%arrange(category,desc(-log10(pval)))
#画图时改变geom_bar的自动排序
data$term<-factor(data$term,levels = unique(data$term),ordered = T)
#作图
ggplot(data)+
geom_bar(aes(x=term,y=-log10(pval),fill=category),stat = 'identity')+
coord_flip()结果:
关于“R语言如何实现柱状图排序和x轴上的标签倾斜操作”这篇文章就分享到这里了,希望以上内容可以对大家有一定的帮助,使各位可以学到更多知识,如果觉得文章不错,请把它分享出去让更多的人看到。
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。