这篇文章将为大家详细讲解有关如何使用Python和Keras进行血管分割,小编觉得挺实用的,因此分享给大家做个参考,希望大家阅读完这篇文章后可以有所收获。
动机:
医学图像的自动分割是提取有用信息的重要步骤,可以帮助医生进行诊断。例如它可以用于分割视网膜血管,可以代表它们的结构并测量它们的宽度,从而可以帮助诊断视网膜疾病。
在这篇文章中,将实现一个神经基线,将图像分割应用于视网膜血管图像。
数据集:
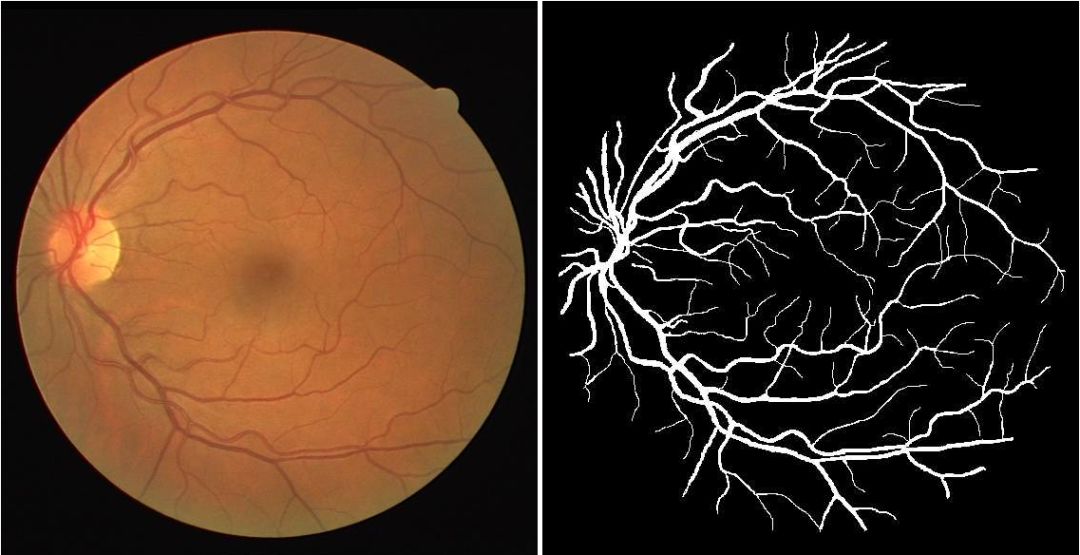
在整个文章中使用DRIVE(数字视网膜图像用于血管提取)数据集进行所有实验。它是40个视网膜图像(20个用于训练,20个用于测试)的数据集,其中血管在像素级注释(参见上面的示例)以标记每个血管的存在(1)或不存在(0)。图像的像素(i,j)。
http://www.isi.uu.nl/Research/Databases/DRIVE/
问题设定:
问题:如果像素是图像中血管的一部分,希望为每个像素分配“1”标签,否则为“0”。
直觉 / 假设:相邻像素值对于对每个像素(i,j)进行预测很重要,因此应该考虑上下文。预测不依赖于图像上的特定位置,因此分类器应具有一些平移不变性。
解决方案:使用CNN!将使用U-net架构进行血管分割。它是一种广泛用于语义分割任务的体系结构,尤其是在医学领域。
型号:
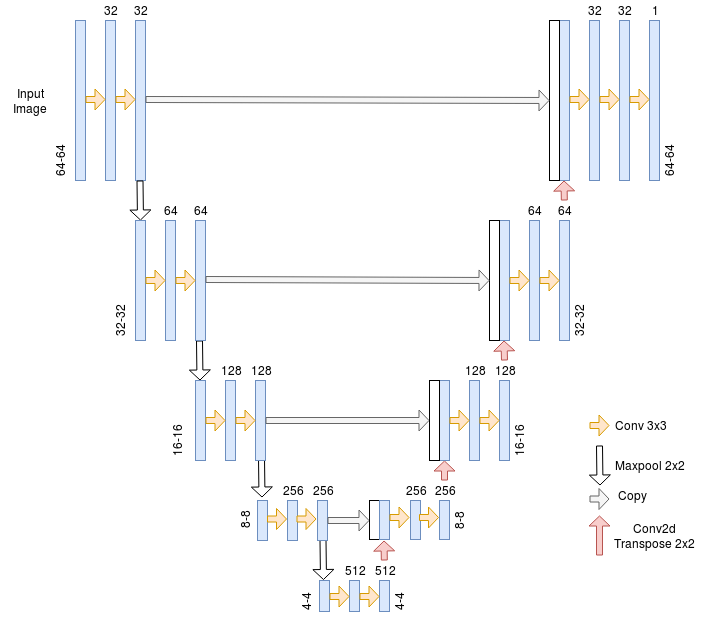
U-Net
U-net架构是编码器 - 解码器,在编码器和解码器之间具有一些跳过连接。该架构的主要优点是能够在对像素进行预测时考虑更广泛的上下文。这要归功于上采样操作中使用的大量通道。
输入图像处理:
在将其反馈到CNN之前应用这一系列处理步骤。
归一化:将像素强度除以255,因此它们在0-1范围内。
裁剪:由于汇集操作,网络期望输入图像的每个维度可被2整除,因此从每个图像中随机裁剪64 * 64。
数据增强:随机翻转(水平或垂直或两者),随机剪切,随机平移(水平或垂直或两者),随机缩放。仅在训练期间执行。
训练三种不同的模型:
预先训练ImageNet VGG编码器+数据增强。
从头开始训练+数据扩充。
从头开始训练而不增加数据。
将使用AUC ROC度量比较这三个模型,将仅在评估中考虑视网膜掩模内的像素(意味着图像圆周围的黑色边缘将不计算)。
结果:
预先训练的编码器+数据增强AUC ROC:0.9820
从头开始训练+数据增加AUC ROC:0.9806
从头开始训练而不增加AUC ROC:0.9811
三种变化的性能接近,但在这种情况下,预训练似乎没有帮助,而数据增加有一点点。
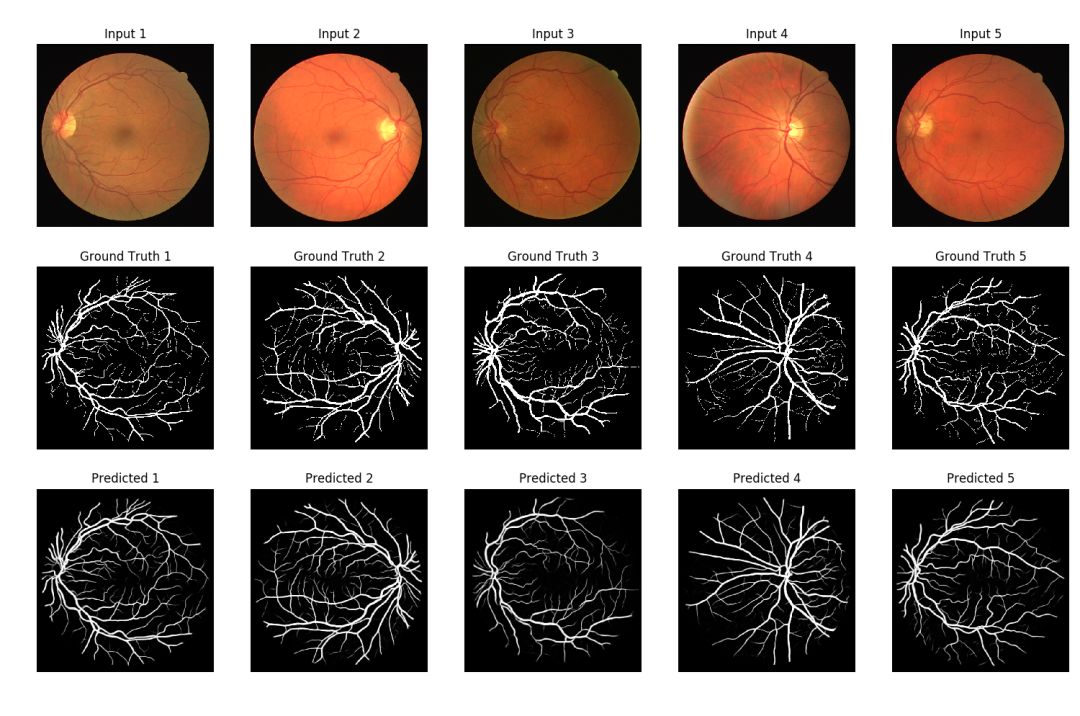
最佳模型预测
上图中的预测看起来很酷!
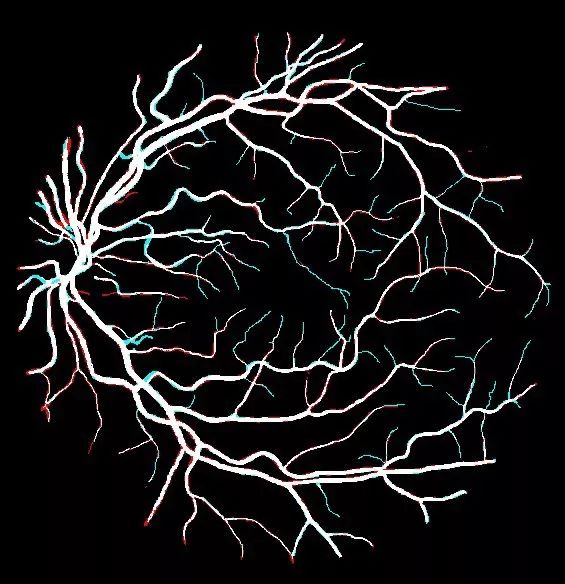
在基本事实之上的预测
还绘制了预测和基本事实之间的差异:蓝色的假阴性和红色的假阳性。可以看到该模型在预测仅一或两个像素宽的细血管方面存在一些困难。
关于“如何使用Python和Keras进行血管分割”这篇文章就分享到这里了,希望以上内容可以对大家有一定的帮助,使各位可以学到更多知识,如果觉得文章不错,请把它分享出去让更多的人看到。
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/3267804/blog/4564797