这篇文章将为大家详细讲解有关eggNOG数据库有什么用,小编觉得挺实用的,因此分享给大家做个参考,希望大家阅读完这篇文章后可以有所收获。
直系同源蛋白的预测在系统发育,比较基因组学等多个领域都占用重要地位,COG数据库开创了同源蛋白数据库的先河,后续又不断有新的数据库涌现,而eggNOG就是目前使用最广泛的数据库之一。
官网如下
http://eggnogdb.embl.de/#/app/home
eggNOG全称evolutionary genealogy of genes:Non-supervised Orthologous, 在COG算法的基础上进行了拓展和延伸,采用基于图状结构的非监督聚类算法,构建了真核,原核,病毒等不同物种的同源蛋白簇。
该数据库最新版本为eggNOG 4.5.1, 涵盖了2031种真核和原核生物,352种病毒,构建了19万个同源蛋白簇。
和COG类似,eggNOG对于orthology group的功能也进行了分类整理,每个类别用一个字母表示,在以下链接可以查看具体的分类信息
http://eggnogdb.embl.de/download/eggnog_4.5/COG_functional_categories.txt
COG数据库中提供了26种分类,eggNOG中提供了25种分类,缺少了如下类别
X Mobilome: prophages, transposons
其他的分类和COG数据库完全一致。在官网的搜索框中可以对OG编号进行检索

COG5157的检索结果如下

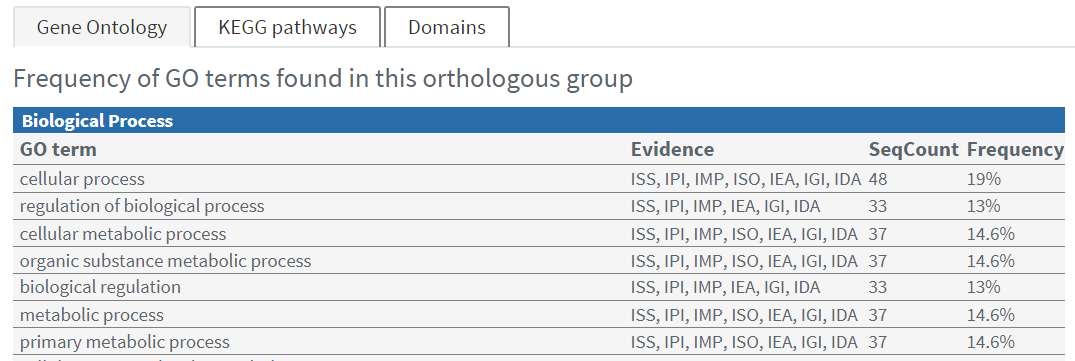
在检索结果中,可以查看该orthology group下包含的蛋白序列和物种信息,也可以下载对应的序列。除此之外,还提供了GO, KEGG, pfam, SMRT等相关的功能注释信息。
除了在线检索外,官方还提供了API服务,方便程序抓取对应数据,示例如下
http://eggnogapi.embl.de/nog_data/text/fasta/ENOG410ZSWV
通过以上API链接,可以获得ENOG410ZSWV这个orthology group下的所有蛋白序列,更多用法请参考官方文档。
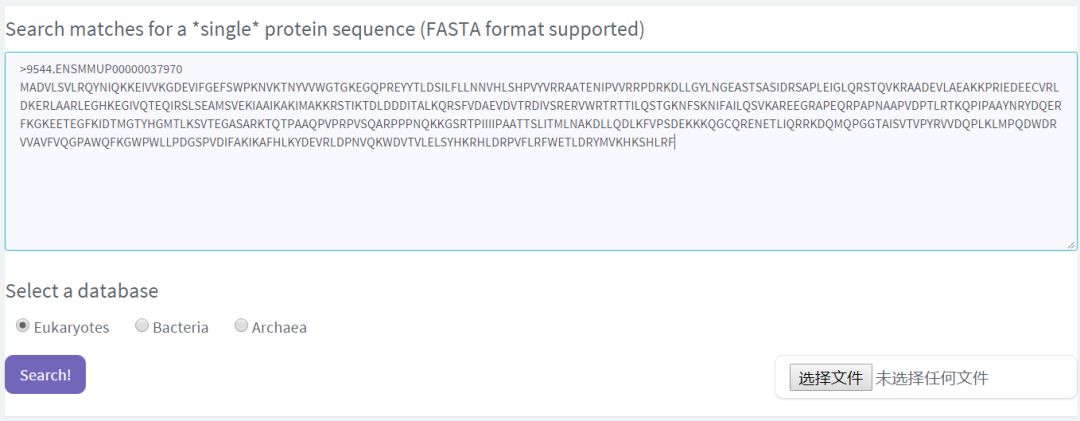
官网还提供了以下两种注释服务
一次只支持一条序列的查询,输入fasta格式的蛋白序列,选择对应物种即可
支持多条序列的NOG注释,提供了在线服务,也可以下载软件到本地运行。
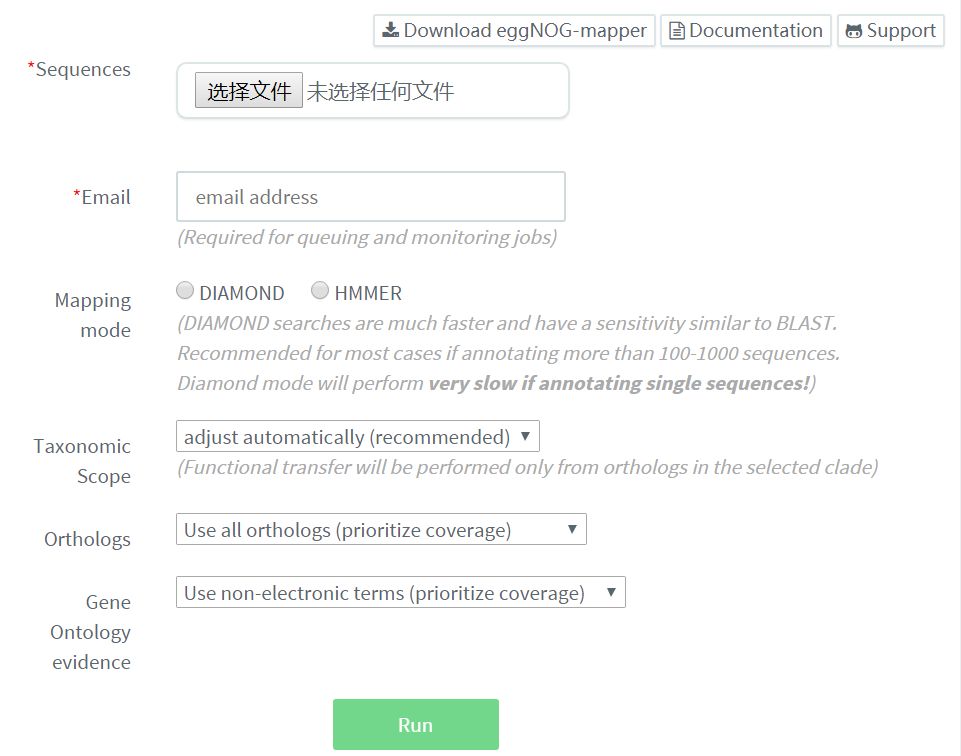
支持diamond和hmmer两种比对软件,当序列条数少于1000条时,选择hmmer,当序列多余1000条时,选择diamond, 速度更快。本地版的eggNOG-mapper 的安装和使用可以参考如下文档
https://github.com/jhcepas/eggnog-mapper/wiki
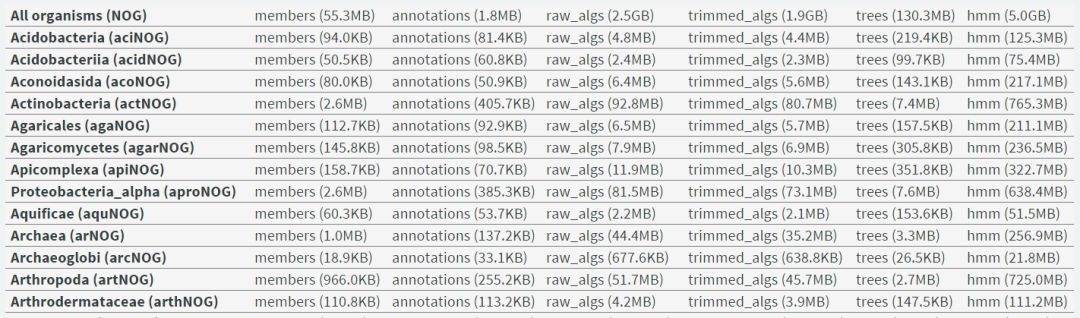
官网提供了下载服务,可以下载完整数据库,也可以根据物种挑选合适的子数据库进行下载,示意如下
通过eggNOG数据库,我们可以对新的蛋白序列进行同源蛋白的注释, 挖掘其功能。
关于“eggNOG数据库有什么用”这篇文章就分享到这里了,希望以上内容可以对大家有一定的帮助,使各位可以学到更多知识,如果觉得文章不错,请把它分享出去让更多的人看到。
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。