本篇内容介绍了“如何使用OncoLnc进行TCGA生存”的有关知识,在实际案例的操作过程中,不少人都会遇到这样的困境,接下来就让小编带领大家学习一下如何处理这些情况吧!希望大家仔细阅读,能够学有所成!
通过收集整理TCGA中不同肿瘤患者的生存数据和基因表达谱信息,OncoLnc提供了一个生存分析的web服务,对应文章的链接如下
https://peerj.com/articles/cs-67/
网址如下
http://www.oncolnc.org/
OncoLnc收集了TCGA中21种肿瘤,共8647个病人的生存数据,以及对应的mRNA和miRNA的表达谱数据。同时收集了来自MiTranscriptome项目lncRNA表达量数据,从而提供了包含mRNA,miRNA,lncRNA 3中基因的生存分析,可以方便的挖掘各种肿瘤中和生存相关的基因。
用法非常的简单,示意如下
在首页的搜索框中输入需要查询的基因,可以是entrez ID, 也可以是gene symbol。以TP53为例,示意如下
点击submit按钮,可以看到事先用cox回归计算好的生存分析结果,示意如下
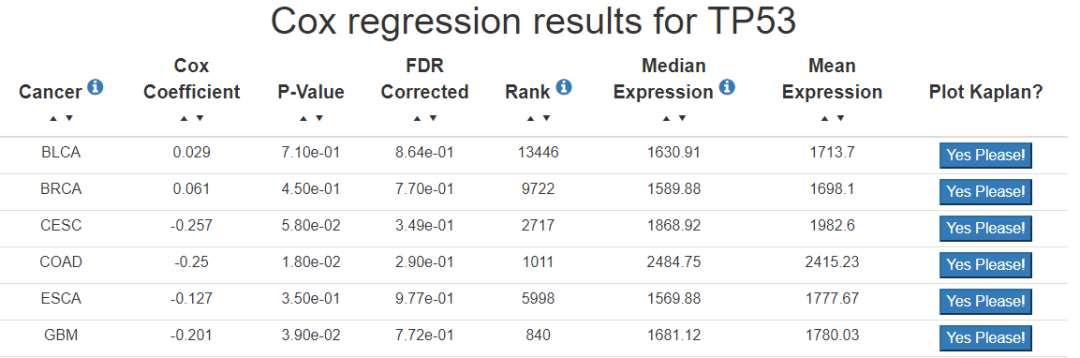
选择感兴趣的肿瘤,可以进一步进行KM生存分析。
为了探究基因和生存的相关性,这里根据基因表达量的大小将样本分为high和low两组,自己指定每组的比例,加起来是100%, 示意如下
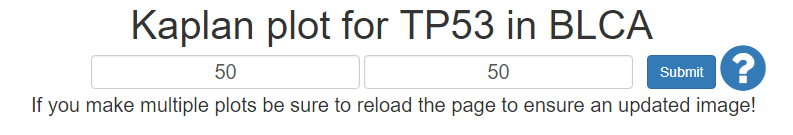
上图中设置两组的比例都是50%,根据该基因的表达量从低到高排序,前50% 定义为地表达组,后50%定义为高表达组,示意如下
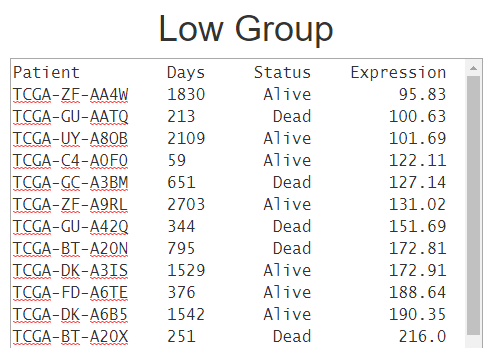
确定好分组之后,点击submit, 就可以得到如下所示的生存分析结果
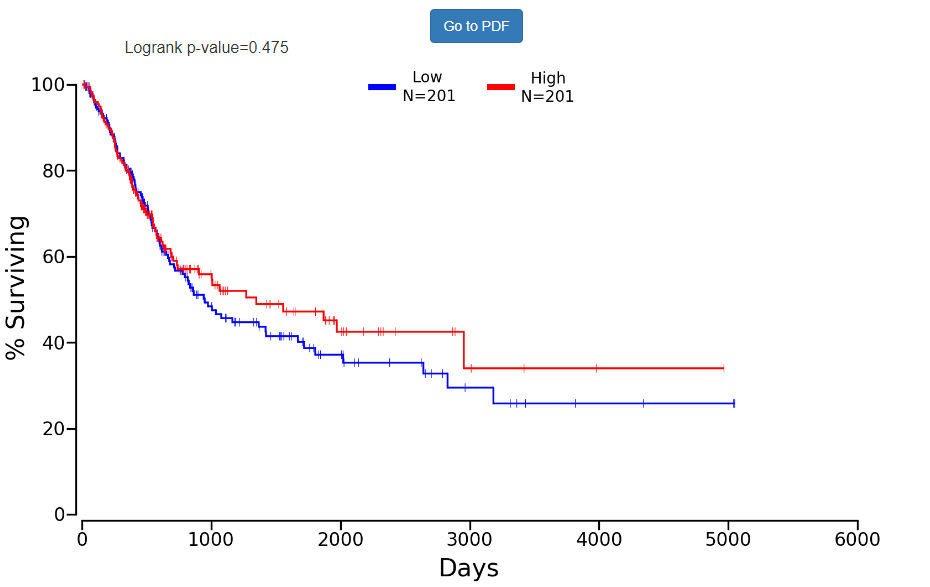
通过OncoLnc可以方便的进行TCGA数据的生存分析,快速的查看多种肿瘤中与生存相关的基因。
“如何使用OncoLnc进行TCGA生存”的内容就介绍到这里了,感谢大家的阅读。如果想了解更多行业相关的知识可以关注亿速云网站,小编将为大家输出更多高质量的实用文章!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4580290/blog/4570482