如何使用bedtools根据染色体上的起止位置拿到基因symbol,相信很多没有经验的人对此束手无策,为此本文总结了问题出现的原因和解决方法,通过这篇文章希望你能解决这个问题。
第一步:将你的染色体位置坐标文件整理成bed格式。
bed格式文件至少包括前3列,分别是:染色体的名字、染色体上的起始位置、染色体上的终止位置。这一步无论用写字板、excel、R等进行处理都可以,文件的后缀名也不重要,因为强行将文件后缀改为bed时,在后面的Linux系统中进行bedtools处理时也会报错。所需的bed格式文件参见下图。
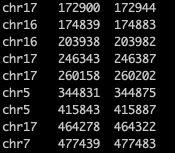
可从gencode中根据自己的需要下载hg38或者hg19版本的人类基因组注释文件(文章中以hg38为例)。这一步可以进gencode官网(https://www.gencodegenes.org/human/)进行本地下载,然后用filezilla等文件传输工具将下载的本地文件传输到服务器。也可以直接在服务器的Linux系统中进行ftp下载。
本地下载:

ftp下载:

获得下载链接后,在Linux系统中输入下面的代码进行ftp下载:
wget ftp://ftp.ebi.ac.uk/pub/databases/gencode/Gencode_human/release_34/gencode.v34.annotation.gtf.gz
在Linux系统中输入下面的代码,得到hg38版本的人类蛋白编码基因的位置坐标:
zcat gencode.v34.annotation.gtf.gz | grep protein_coding |perl -alne '{next unless $F[2] eq "gene" ;/gene_name \"(.*?)\";/; print "$F[0]\t$F[3]\t$F[4]\t$1" }' >protein_coding.hg38.position先将待处理的坐标bed格式文件链接或复制到第三步得到的结果文件所在的目录下,然后修改这一文件的后缀名为bed,再将这一文件转化为Tab键分隔的后缀名为bed的文件,需输入下面的代码(motif1.bed是自己命名的待处理坐标文件):
mv motif1.tsv motif1.bedperl -p -i -e 's/ /\t/g' motif1.bed如果在第一步的时候已将待处理的bed格式文件保存为了Tab键分隔格式,但是在后面的处理中仍然报错,不妨再进行一次Tab键分隔处理。
首先需要启动自己安装了bedtools软件的conda小环境,然后输入下面的代码:
bedtools intersect -a motif1.bed -b ~/dna/exercise/protein_coding.hg38.position -wa -wb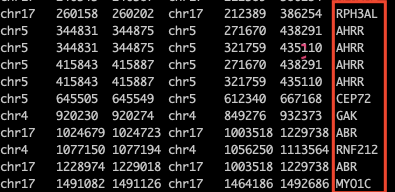
也可以对结果进行汇总,将位于相同染色体坐标的基因symbol写在一块,此时只需要加上|后面的代码即可。| 之前的文件得到的结果有几列,-c后面的数字就写几。如我得到的有7列,-c后面就写7。
bedtools intersect -a motif1.bed -b ~/dna/exercise/protein_coding.hg38.position -wa -wb | bedtools groupby -i - -g 1-4 -c 7 -o collapse
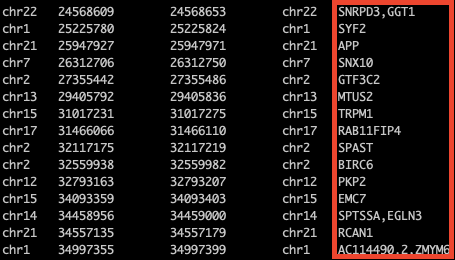
也可以另存结果:
bedtools intersect -a motif1.bed -b ~/dna/exercise/protein_coding.hg38.position -wa -wb | bedtools groupby -i - -g 1-4 -c 7 -o collapse >gene.tsv
新保存的gene.tsv文件就是结果文件了,然后可以拿着结果进行后续处理啦~。
看完上述内容,你们掌握如何使用bedtools根据染色体上的起止位置拿到基因symbol的方法了吗?如果还想学到更多技能或想了解更多相关内容,欢迎关注亿速云行业资讯频道,感谢各位的阅读!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4503882/blog/4352287