这期内容当中小编将会给大家带来有关如何分析GDC数据库中的数据的R语言包GDC RNATools,文章内容丰富且以专业的角度为大家分析和叙述,阅读完这篇文章希望大家可以有所收获。
GDCRNATools:加利福尼亚大学生物与植物科学系植物基因组学中LNCRNA、miRNA和mRNA数据的综合分析软件包
GDC:基因组数据共享
帮助文档链接 http://bioconductor.org/packages/devel/bioc/vignettes/GDCRNATools/inst/doc/GDCRNATools.html
library(GDCRNATools)
project<-'TCGA-CHOL'
rnadir<-paste(project,'RNAseq',sep='/')
mirdir<-paste(project,'miRNAs',sep="/")
gdcRNADownload(project.id = 'TCGA-CHOL',
data.type = 'RNAseq',
write.manifest = F,
method = 'gdc-client',
directory = rnadir)
在linux系统中重复到这一步的时候遇到报错 ImportError: /lib64/libc.so.6: version `GLIBC_2.18' not found (required by /tmp/_MEIylVP0W/libstdc++
我的解决办法是把它默认下载的gdc-client_v1.3.0替换掉,我换成gdc-client_v1.5.0,下载地址是https://gdc.cancer.gov/access-data/gdc-data-transfer-tool
gdcRNADownload(project.id = 'TCGA-CHOL',
data.type = 'miRNAs',
write.manifest = F,
method = 'gdc-client',
directory = mirdir)
clinicaldir<-paste(project,'Clinical',sep='/')
gdcClinicalDownload(project.id = 'TCGA-CHOL',
write.manifest = F,
method='gdc-client',
directory = clinicaldir)
metaMatrix.RNA<-gdcParseMetadata(project.id = 'TCGA-CHOL',
data.type = 'RNAseq',
write.meta = F)
metaMatrix.RNA<-gdcFilterDuplicate(metaMatrix.RNA)
metaMatrix.RNA<-gdcFilterSampleType(metaMatrix.RNA)
metaMatrix.MIR<-gdcParseMetadata(project.id = 'TCGA-CHOL',
data.type = 'miRNAs',
write.meta = F)
metaMatrix.MIR
metaMatrix.MIR<-gdcFilterDuplicate(metaMatrix.MIR)
metaMatrix.MIR<-gdcFilterSampleType(metaMatrix.MIR)
rnaCounts<-gdcRNAMerge(metadata = metaMatrix.RNA,
path = rnadir,
organized = FALSE,
data.type = 'RNAseq')
mirCounts<-gdcRNAMerge(metadata = metaMatrix.MIR,
path = mirdir,
organized = FALSE,
rnaCounts[1:5,1:5]
mirCounts[1:5,1:5]
rnaExpr<-gdcVoomNormalization(counts=rnaCounts,filter=F)
mirExpr<-gdcVoomNormalization(counts=mirCounts,filter=F)
rnaExpr[1:5,1:5]
mirExpr[1:5,1:5]
DEGAll<-gdcDEAnalysis(counts = rnaCounts,
group=metaMatrix.RNA$sample_type,
comparison = 'PrimaryTumor-SolidTissueNormal',
method='limma')
deALL<-gdcDEReport(deg=DEGAll,gene.type = 'all')
deLNC<-gdcDEReport(deg=DEGAll,gene.type='long_non_coding')
dePC<-gdcDEReport(deg=DEGAll,gene.type = 'protein_coding')
gdcBarPlot(deg=deALL,angle = 45,data.type = 'RNAseq')
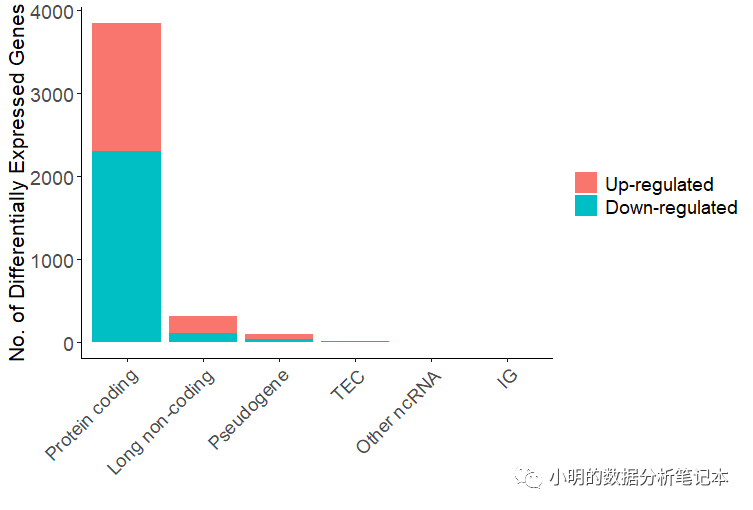
这里TEC和IG分别是啥?
gdcVolcanoPlot(deLNC)
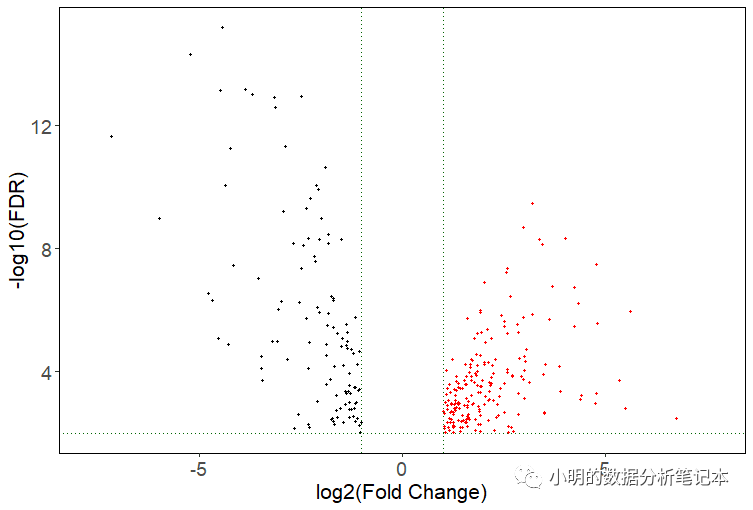
degName<-rownames(deLNC)
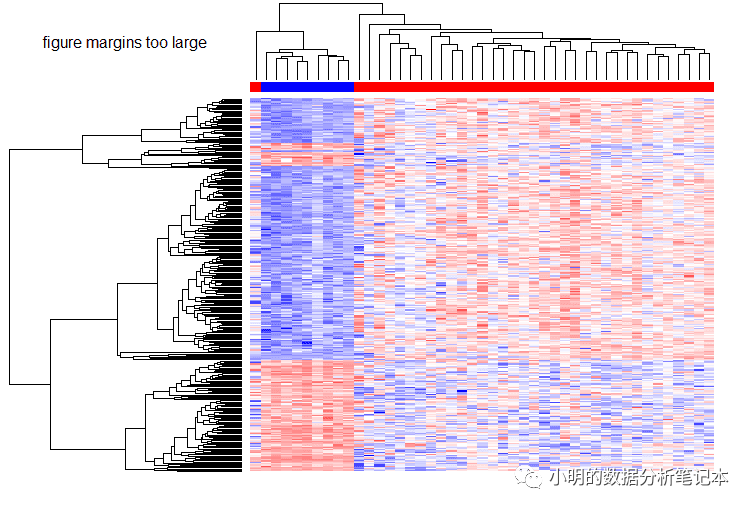
gdcHeatmap(deg.id = degName,metadata = metaMatrix.RNA,rna.expr = rnaExpr)
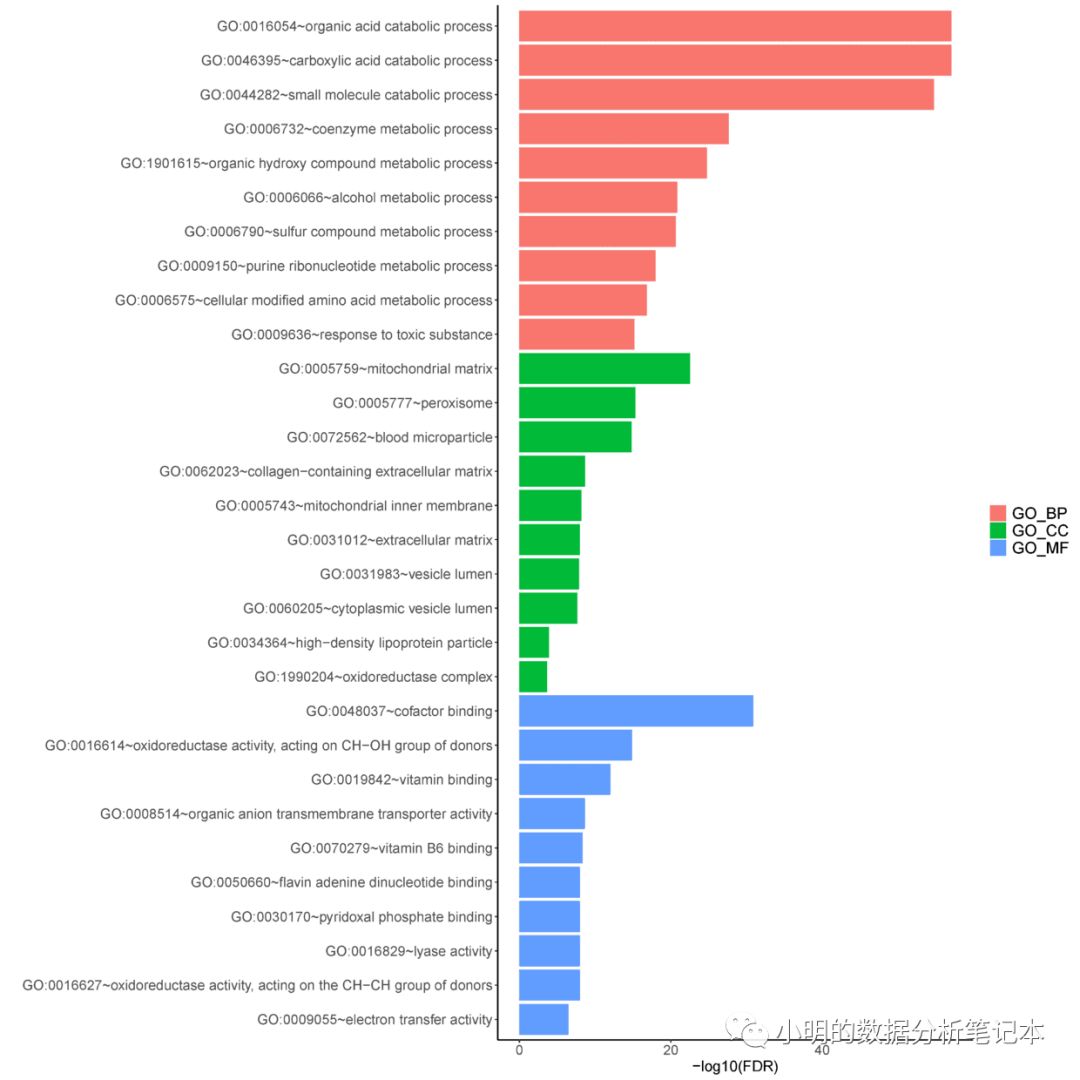
enrichOutput<-gdcEnrichAnalysis(gene=rownames(deALL),
simplify=T)
gdcEnrichPlot(enrichOutput,type='bar',category = 'GO',num.terms = 10)
画图的时候遇到报错 Error in .Call.graphics(C_palette2, .Call(C_palette2, NULL)) : invalid graphics state 不知道原因出在哪里,但是保存到本地没问题
pdf(file="../goenrich.pdf",width = 15,height = 15)
gdcEnrichPlot(enrichOutput,type='bar',category = 'GO',num.terms = 10)
dev.off()
ceOUtput<-gdcCEAnalysis(lnc=rownames(deLNC),
pc=rownames(dePC),
lnc.targets = 'starBase',
pc.targets = 'starBase',
rna.expr = rnaExpr,
mir.expr = mirExpr)
edges<-gdcExportNetwork(ceNetwork = ceOutput2,net='edges')
nodes<-gdcExportNetwork(ceNetwork = ceOutput2,net='nodes')
write.table(edges,file='edges.txt',sep='\t',quote=F)
write.table(nodes,file="nodes.txt",sep="\t",quote=F)最后生成了两个文件,如何用cytoscape可视化这两个文件我暂时还不知道如何实现。
上述就是小编为大家分享的如何分析GDC数据库中的数据的R语言包GDC RNATools了,如果刚好有类似的疑惑,不妨参照上述分析进行理解。如果想知道更多相关知识,欢迎关注亿速云行业资讯频道。
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。