这篇文章主要介绍使用Python实现KMeans聚类算法的案例,文中介绍的非常详细,具有一定的参考价值,感兴趣的小伙伴们一定要看完!
一 、关于初始聚类中心的选取
初始聚类中心的选择一般有:
(1)随机选取
(2)随机选取样本中一个点作为中心点,在通过这个点选取距离其较大的点作为第二个中心点,以此类推。
(3)使用层次聚类等算法更新出初始聚类中心
我一开始是使用numpy随机产生k个聚类中心
Center = np.random.randn(k,n)
但是发现聚类的时候迭代几次以后聚类中心会出现nan,有点搞不清楚怎么回事
所以我分别尝试了:
(1)选择数据集的前K个样本做初始中心点
(2)选择随机K个样本点作为初始聚类中心
发现两者都可以完成聚类,我是用的是iris.csv数据集,在选择前K个样本点做数据集时,迭代次数是固定的,选择随机K个点时,迭代次数和随机种子的选取有关,而且聚类效果也不同,有的随机种子聚类快且好,有的慢且差。
def InitCenter(k,m,x_train): #Center = np.random.randn(k,n) #Center = np.array(x_train.iloc[0:k,:]) #取数据集中前k个点作为初始中心 Center = np.zeros([k,n]) #从样本中随机取k个点做初始聚类中心 np.random.seed(5) #设置随机数种子 for i in range(k): x = np.random.randint(m) Center[i] = np.array(x_train.iloc[x]) return Center
二 、关于类间距离的选取
为了简单,我直接采用了欧氏距离,目前还没有尝试其他的距离算法。
def GetDistense(x_train, k, m, Center): Distence=[] for j in range(k): for i in range(m): x = np.array(x_train.iloc[i, :]) a = x.T - Center[j] Dist = np.sqrt(np.sum(np.square(a))) # dist = np.linalg.norm(x.T - Center) Distence.append(Dist) Dis_array = np.array(Distence).reshape(k,m) return Dis_array
三 、关于终止聚类条件的选取
关于聚类的终止条件有很多选择方法:
(1)迭代一定次数
(2)聚类中心的更新小于某个给定的阈值
(3)类中的样本不再变化
我用的是前两种方法,第一种很简单,但是聚类效果不好控制,针对不同数据集,稳健性也不够。第二种比较合适,稳健性也强。第三种方法我还没有尝试,以后可以试着用一下,可能聚类精度会更高一点。
def KMcluster(x_train,k,n,m,threshold):
global axis_x, axis_y
center = InitCenter(k,m,x_train)
initcenter = center
centerChanged = True
t=0
while centerChanged:
Dis_array = GetDistense(x_train, k, m, center)
center ,axis_x,axis_y,axis_z= GetNewCenter(x_train,k,n,Dis_array)
err = np.linalg.norm(initcenter[-k:] - center)
print(err)
t+=1
plt.figure(1)
p=plt.subplot(3, 3, t)
p1,p2,p3 = plt.scatter(axis_x[0], axis_y[0], c='r'),plt.scatter(axis_x[1], axis_y[1], c='g'),plt.scatter(axis_x[2], axis_y[2], c='b')
plt.legend(handles=[p1, p2, p3], labels=['0', '1', '2'], loc='best')
p.set_title('Iteration'+ str(t))
if err < threshold:
centerChanged = False
else:
initcenter = np.concatenate((initcenter, center), axis=0)
plt.show()
return center, axis_x, axis_y,axis_z, initcentererr是本次聚类中心点和上次聚类中心点之间的欧氏距离。
threshold是人为设定的终止聚类的阈值,我个人一般设置为0.1或者0.01。
为了将每次迭代产生的类别显示出来我修改了上述代码,使用matplotlib展示每次迭代的散点图。
下面附上我测试数据时的图,子图设置的个数要根据迭代次数来定。
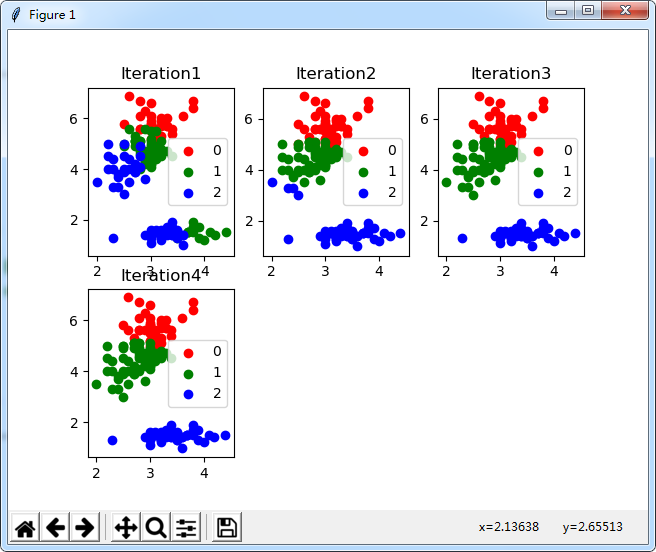
我测试了几个数据集,聚类的精度还是可以的。
使用iris数据集分析的结果为:
err of Iteration 1 is 3.11443180281
err of Iteration 2 is 1.27568813621
err of Iteration 3 is 0.198909381512
err of Iteration 4 is 0.0
Final cluster center is [[ 6.85 3.07368421 5.74210526 2.07105263]
[ 5.9016129 2.7483871 4.39354839 1.43387097]
[ 5.006 3.428 1.462 0.246 ]]
最后附上全部代码,错误之处还请多多批评,谢谢。
#encoding:utf-8
"""
Author: njulpy
Version: 1.0
Data: 2018/04/11
Project: Using Python to Implement KMeans Clustering Algorithm
"""
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from sklearn.cluster import KMeans
def InitCenter(k,m,x_train):
#Center = np.random.randn(k,n)
#Center = np.array(x_train.iloc[0:k,:]) #取数据集中前k个点作为初始中心
Center = np.zeros([k,n]) #从样本中随机取k个点做初始聚类中心
np.random.seed(15) #设置随机数种子
for i in range(k):
x = np.random.randint(m)
Center[i] = np.array(x_train.iloc[x])
return Center
def GetDistense(x_train, k, m, Center):
Distence=[]
for j in range(k):
for i in range(m):
x = np.array(x_train.iloc[i, :])
a = x.T - Center[j]
Dist = np.sqrt(np.sum(np.square(a))) # dist = np.linalg.norm(x.T - Center)
Distence.append(Dist)
Dis_array = np.array(Distence).reshape(k,m)
return Dis_array
def GetNewCenter(x_train,k,n, Dis_array):
cen = []
axisx ,axisy,axisz= [],[],[]
cls = np.argmin(Dis_array, axis=0)
for i in range(k):
train_i=x_train.loc[cls == i]
xx,yy,zz = list(train_i.iloc[:,1]),list(train_i.iloc[:,2]),list(train_i.iloc[:,3])
axisx.append(xx)
axisy.append(yy)
axisz.append(zz)
meanC = np.mean(train_i,axis=0)
cen.append(meanC)
newcent = np.array(cen).reshape(k,n)
NewCent=np.nan_to_num(newcent)
return NewCent,axisx,axisy,axisz
def KMcluster(x_train,k,n,m,threshold):
global axis_x, axis_y
center = InitCenter(k,m,x_train)
initcenter = center
centerChanged = True
t=0
while centerChanged:
Dis_array = GetDistense(x_train, k, m, center)
center ,axis_x,axis_y,axis_z= GetNewCenter(x_train,k,n,Dis_array)
err = np.linalg.norm(initcenter[-k:] - center)
t+=1
print('err of Iteration '+str(t),'is',err)
plt.figure(1)
p=plt.subplot(2, 3, t)
p1,p2,p3 = plt.scatter(axis_x[0], axis_y[0], c='r'),plt.scatter(axis_x[1], axis_y[1], c='g'),plt.scatter(axis_x[2], axis_y[2], c='b')
plt.legend(handles=[p1, p2, p3], labels=['0', '1', '2'], loc='best')
p.set_title('Iteration'+ str(t))
if err < threshold:
centerChanged = False
else:
initcenter = np.concatenate((initcenter, center), axis=0)
plt.show()
return center, axis_x, axis_y,axis_z, initcenter
if __name__=="__main__":
#x=pd.read_csv("8.Advertising.csv") # 两组测试数据
#x=pd.read_table("14.bipartition.txt")
x=pd.read_csv("iris.csv")
x_train=x.iloc[:,1:5]
m,n = np.shape(x_train)
k = 3
threshold = 0.1
km,ax,ay,az,ddd = KMcluster(x_train, k, n, m, threshold)
print('Final cluster center is ', km)
#2-Dplot
plt.figure(2)
plt.scatter(km[0,1],km[0,2],c = 'r',s = 550,marker='x')
plt.scatter(km[1,1],km[1,2],c = 'g',s = 550,marker='x')
plt.scatter(km[2,1],km[2,2],c = 'b',s = 550,marker='x')
p1, p2, p3 = plt.scatter(axis_x[0], axis_y[0], c='r'), plt.scatter(axis_x[1], axis_y[1], c='g'), plt.scatter(axis_x[2], axis_y[2], c='b')
plt.legend(handles=[p1, p2, p3], labels=['0', '1', '2'], loc='best')
plt.title('2-D scatter')
plt.show()
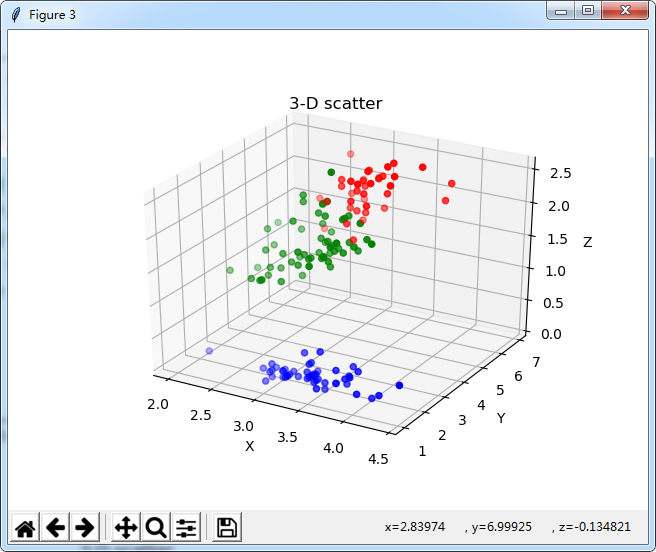
#3-Dplot
plt.figure(3)
TreeD = plt.subplot(111, projection='3d')
TreeD.scatter(ax[0],ay[0],az[0],c='r')
TreeD.scatter(ax[1],ay[1],az[1],c='g')
TreeD.scatter(ax[2],ay[2],az[2],c='b')
TreeD.set_zlabel('Z') # 坐标轴
TreeD.set_ylabel('Y')
TreeD.set_xlabel('X')
TreeD.set_title('3-D scatter')
plt.show()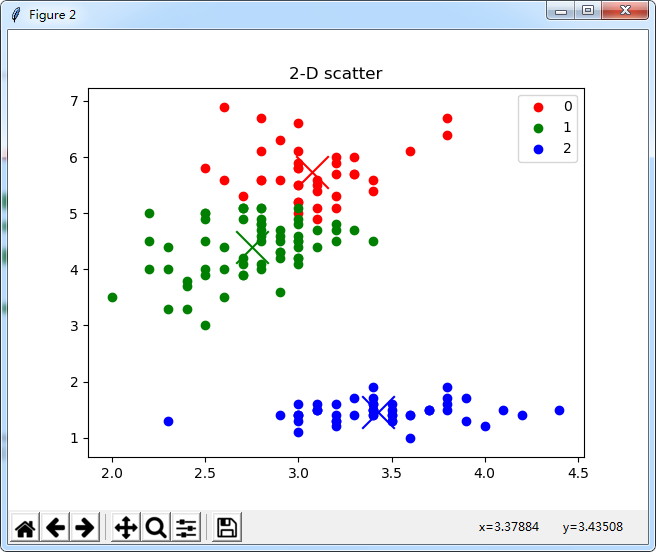
以上是“使用Python实现KMeans聚类算法的案例”这篇文章的所有内容,感谢各位的阅读!希望分享的内容对大家有帮助,更多相关知识,欢迎关注亿速云行业资讯频道!
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。