这篇文章主要介绍了PLEK工具有什么用,具有一定借鉴价值,感兴趣的朋友可以参考下,希望大家阅读完这篇文章之后大有收获,下面让小编带着大家一起了解一下。
在之前的文章中,我们介绍过CPC和CNCI这两款软件,可以用于预测lncRNA序列。其中CPC基于序列比对的方式,对于注释信息相对全面的物种分类效果较好,但是运行速度相对较慢,CNCI基于序列的三联体碱基组成来区分编码和非编码转录本,对于注释信息缺乏的物种,效果也不错,但是当序列中存在插入缺失时,其分类效果就变得很差。
在高通量测序产生的数据中,会存在一定的测序错误,虽然比例很低,但是基于这样的序列组装得到转录本然后去预测lncRNA, 对于CNCI这个软件而言,就会造成相当大的影响。
为了克服上述问题,需要一款运行速度又快,又可以一定程度上降低测序错误影响的lncRNA预测软件,PLEK软件就是基于这样的出发点进行开发的。PLEK软件通过序列的kmer构成来区分编码和非编码转录本,不需要通过比对来完成,所以运行速度较快,同时其性能受到测序错误的影响的概率较低,比较稳定。
在论文中,开发者评估了测序错误对各个软件准确度的影响,结果如下所示
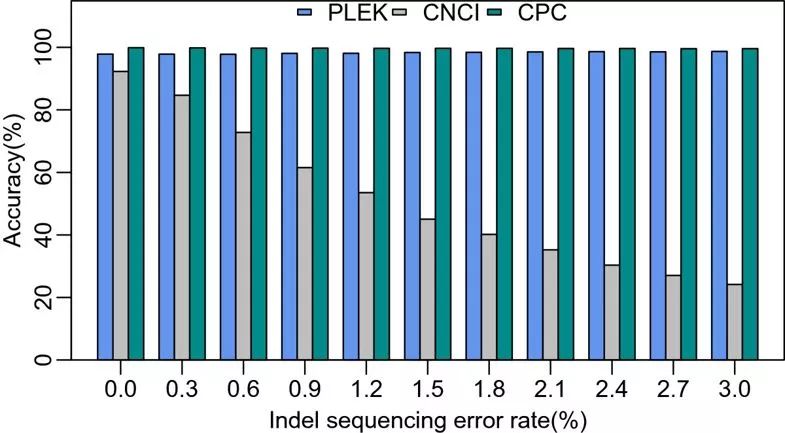
可以看到,随着测序错误比例的上升,CNCI的准确度急剧下降,而PLEK和CPC的结果都相对稳定。
同时利用小鼠的转录本数据,评估了各个软件分类的准确性,结果如下所示
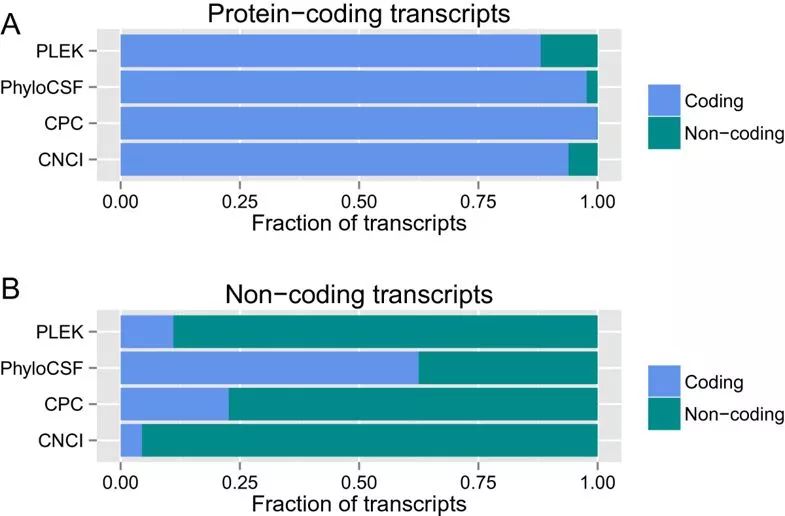
从蛋白编码的转录本来看,CPC的准确率最高,PLEK误判的概率最高;从非编码转录本来看,CNCI的准确率最高,phyloCSF的误判率最高。
综合来看,PLEK的准确性介于CPC和CNCI之间,但是考虑到测序错误的影响,PLEK的优势会更加明显。
论文中对于各个软件的运行效率,也进行了比较,结果如下
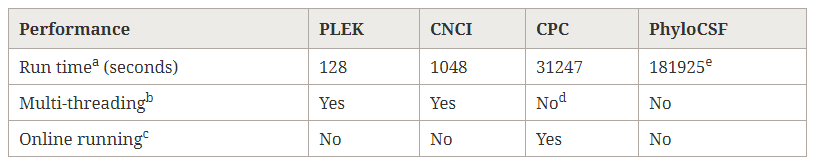
可以看到PLEK的运行速度是最快的。该软件的源代码托管在sourceforge上,网址如下
https://sourceforge.net/projects/plek/files/
安装方式如下
wget https://sourceforge.net/projects/plek/files/PLEK.1.2.tar.gz tar xzvf PLEK.1.2.tar.gz cd PLEK.1.2 python PLEK_setup.py
基本用法如下
python PLEK.py \ -fasta transcript.fa \ -out output \ -thread 10
只需要输入转录本对应的fasta格式的文件就可以了,输出文件output内容示意如下
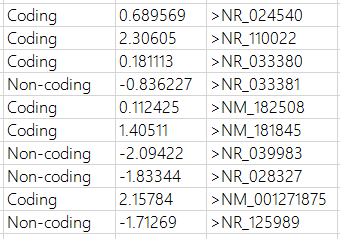
第一列代表该转录本为coding还是non-coding, 第二列为打分值,打分值大于0为coding, 小于零为non-coding, 第三列为fasta文件中的序列标识符。
默认情况下会调用内置的svm模型,如果你有该物种已知的mRNA和lncRNA转录本序列,也可以构建自己的模型,代码如下
python PLEKModelling.py \ -mRNA mRNAs.fa \ -lncRNA lncRNAs.fa \ -prefix 20190129
运行成功后,会生成后缀为.model和.range的两个文件。在预测时可以通过参数指定svm模型,用法如下
python PLEK.py \ -fasta transcript.fa \ -out output \ -model 20190129.model -range 20190129.range \ -thread 10
感谢你能够认真阅读完这篇文章,希望小编分享的“PLEK工具有什么用”这篇文章对大家有帮助,同时也希望大家多多支持亿速云,关注亿速云行业资讯频道,更多相关知识等着你来学习!
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。