本篇内容主要讲解“如何使用phantompeakqualtools进行cross correlation分析”,感兴趣的朋友不妨来看看。本文介绍的方法操作简单快捷,实用性强。下面就让小编来带大家学习“如何使用phantompeakqualtools进行cross correlation分析”吧!
ENCODE官方提供了chip_seq分析的pipeline以供参考,在peak calling前的预处理环节,流程示意如下
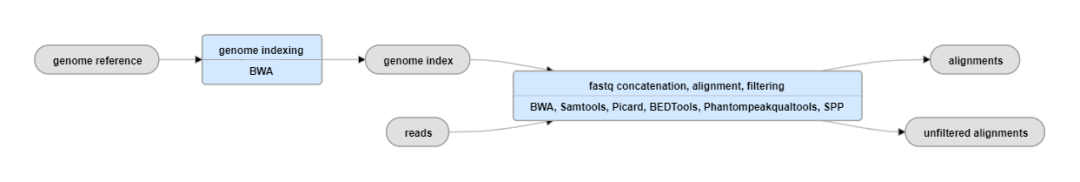
可以看到其中包含了一个名为phantompeakqualtools的工具,这个工具可以进行cross-correlation分析,计算得到NSC和RSC两个指标值,软件的源代码保存在github上,网址如下
https://github.com/kundajelab/phantompeakqualtools
安装过程如下
docker run -it -v /home:/home --rm centos
yum install -y epel-release
yum install -y R
yum install -y wget
yum install -y ncurses-devel bzip2 libcurl libcurl-devel
# samtools
wget https://sourceforge.net/projects/samtools/files/samtools/1.9/samtools-1.9.tar.bz2
tar xjvf samtools-1.9.tar.bz2
cd samtools-1.9
./configure
make
make install
# R
install.packages("BiocManager")
BiocManager::install("Rsamtools", version = "3.8")
install.packages("spp")
install.packages("snow")
install.packages("snowfall")
# phantompeakqualtools
wget https://github.com/kundajelab/phantompeakqualtools/archive/1.2.tar.gz
tar xzvf 1.2.tar.gz基本用法如下
Rscript run_spp.R \
-c=chip.bam \
-rf -p=10 \
-filtchr="chrM" \
-savp=cc.plot.pdf \
-out=cc.score.txt软件的运行时间很长,pdf结果示意如下
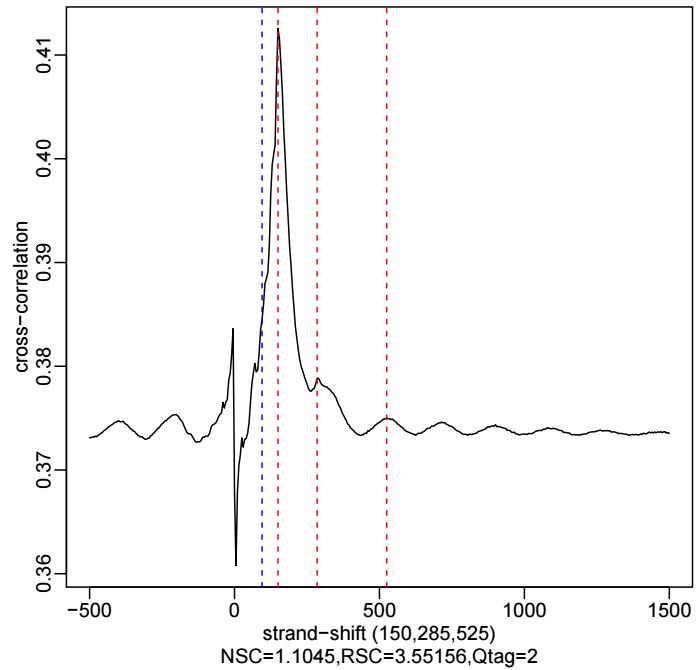
蓝色标记的是phantome peak的位置。在txt输出文件中,每一列代表一个指标,但是没有表头,可以参考输出到屏幕上的log信息来理解,示意如下
Minimum cross-correlation value 0.3735286
Minimum cross-correlation shift 1500
Top 3 cross-correlation values 0.412562930952545,0.37885721605778,0.375020285168966
Top 3 estimates for fragment length 150,285,525
Window half size 200
Phantom peak location 95
Phantom peak Correlation 0.3845194
Normalized Strand cross-correlation coefficient (NSC) 1.104502
Relative Strand cross-correlation Coefficient (RSC) 3.551565
Phantom Peak Quality Tag 2到此,相信大家对“如何使用phantompeakqualtools进行cross correlation分析”有了更深的了解,不妨来实际操作一番吧!这里是亿速云网站,更多相关内容可以进入相关频道进行查询,关注我们,继续学习!
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。