这篇文章主要讲解了“eggnog-mapper软件的安装配置方法”,文中的讲解内容简单清晰,易于学习与理解,下面请大家跟着小编的思路慢慢深入,一起来研究和学习“eggnog-mapper软件的安装配置方法”吧!
数据库文件下载链接
http://eggnogdb.embl.de/download/emapperdb-5.0.0/eggnog.db.gz http://eggnogdb.embl.de/download/emapperdb-5.0.0/eggnog_proteins.dmnd.gz
将数据库文件下载到data文件夹下
这次使用wget命令成功下载,试着运行软件自带数据成功
python emapper.py -i test/test_queries.fa --output out -m diamond --cpu 8这个软件是python2版本的,使用之前我先创建了一个python2的虚拟环境
conda create -n emapper python=2.7conda activate emapper以上是使用diamond做注释,这个软件还可以使用hmmer做注释,印象里好像还得配置hmmer的数据库。而且这个数据库文件非常大,而且数据库文件有好几个,这里我先不尝试了。
同时这个软件还有一个在线版 http://eggnogdb.embl.de/#/app/emapper
在线版运行也很快,我之前试过两万多条蛋白序列,基本上一个晚上就能得到结果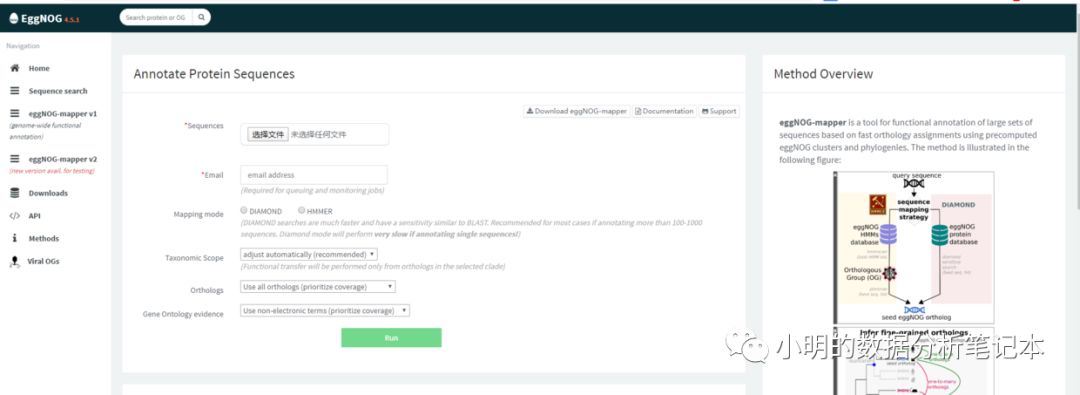
软件的详细介绍
https://blog.csdn.net/woodcorpse/article/details/83144768
感谢各位的阅读,以上就是“eggnog-mapper软件的安装配置方法”的内容了,经过本文的学习后,相信大家对eggnog-mapper软件的安装配置方法这一问题有了更深刻的体会,具体使用情况还需要大家实践验证。这里是亿速云,小编将为大家推送更多相关知识点的文章,欢迎关注!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4579431/blog/4347632