本篇内容介绍了“R语言中ggtree怎么从进化树中挑选子集”的有关知识,在实际案例的操作过程中,不少人都会遇到这样的困境,接下来就让小编带领大家学习一下如何处理这些情况吧!希望大家仔细阅读,能够学有所成!
已经有了100个物种进化树文件,我想从这个树文件里挑选出10个我感兴趣的物种的进化关系。
treeio这个R语言包里有一个函数drop.tip()可以实现,但是他不是直接挑选出来感兴趣的,而是去掉不感兴趣的。
树文件使用treeio包里带的示例文件sample.nwk
nwk<-system.file("extdata","sample.nwk",package="treeio")library(ggtree)tree<-read.tree(nwk)ggtree(tree)+ geom_text2(aes(subset=!isTip,label=node))+ geom_tiplab()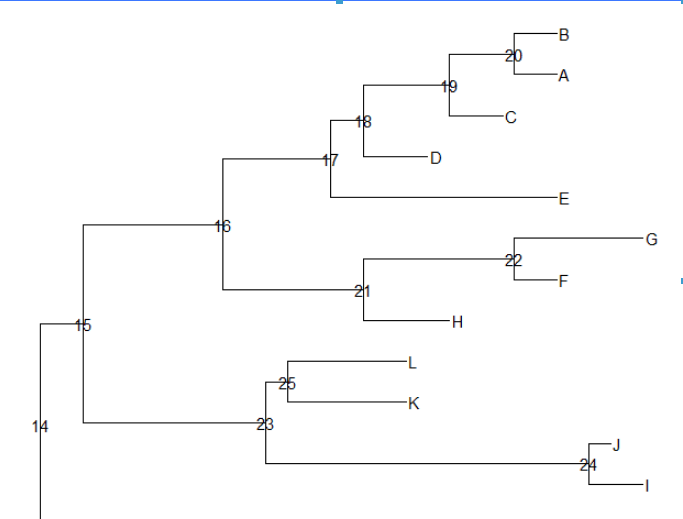
如果我只想看看A/B/C/D/E这些物种之间的关系,就可以去掉非ABCDE这些tips。使用到的是drop.tip()函数。
两个参数:第一个读进来的数;第二个参数是想要去掉的tips。
reserve_tip<-c("A","B","C","D","E")to_drop<-tree$tip.label[-match(reserve_tip,tree$tip.label)]to_droptree_reduced<-treeio::drop.tip(tree,to_drop)ggtree(tree_reduced)+ geom_tiplab()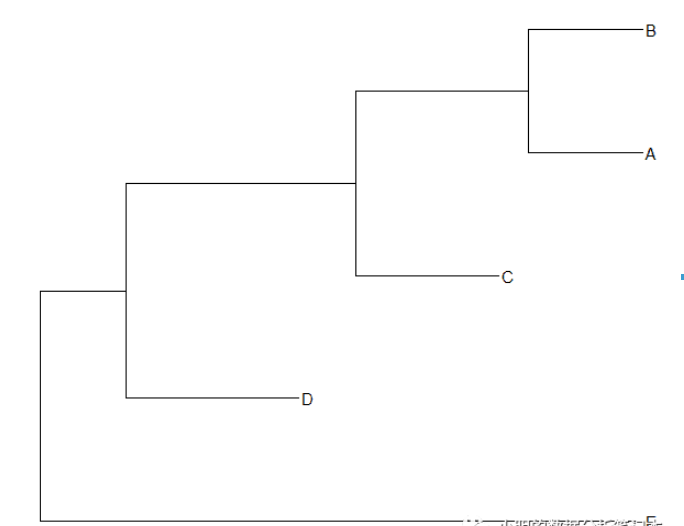
treeio::write.nexus(tree_reduced,file="../../tree_reduced.nex")“R语言中ggtree怎么从进化树中挑选子集”的内容就介绍到这里了,感谢大家的阅读。如果想了解更多行业相关的知识可以关注亿速云网站,小编将为大家输出更多高质量的实用文章!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4579431/blog/4347586