今天就跟大家聊聊有关基于家系数据的GWAS分析是怎样的,可能很多人都不太了解,为了让大家更加了解,小编给大家总结了以下内容,希望大家根据这篇文章可以有所收获。
通过GWAS分析可以寻找与某一疾病或性状相关的突变位点,传统的GWAS都是基于control/case的设计,通过比较健康人群和患病人群中突变位点或者基因型频率的差异,最终确定相关的位点。
对于家族遗传病而言,上述的分析策略就存在问题了。在家系中,不同世代的个体之间存在遗传关系,疾病相关的位点也会有父代传递给子代。为了将这个传递关系考虑进来,针对家族遗传病的GWAS分析,提出了新的分析方法-TDT。
TDT全称 TRANSMISSION DISEQUILIBRIUM TEST,通过分析从父代继承的allel个数和期望的allel个数的差异,从而判断改为点是否与疾病相关。
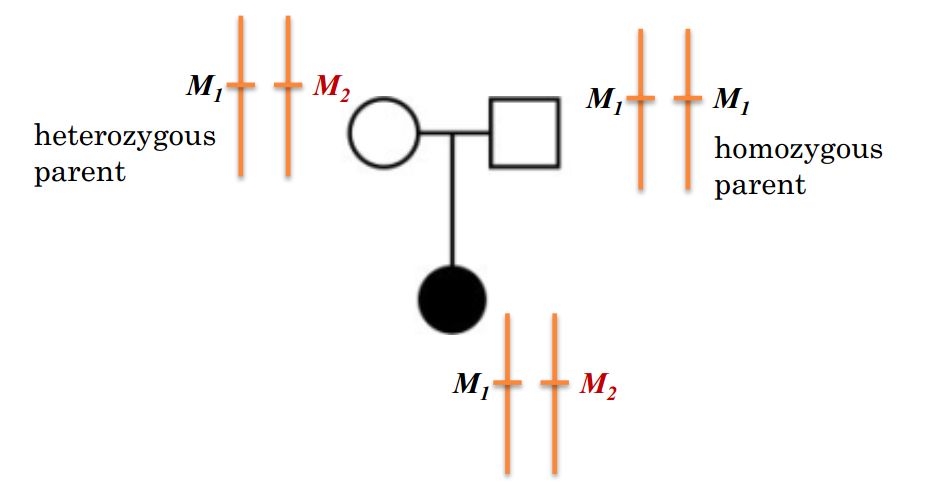
在上述的示意图中,子代从纯合父代继承了M1allel, 从杂合父代继承了M2 allel,由父代传递给子代的allel 就叫做 transmitted allel。
对于一个SNP位点而言,统计样本中transmitted allel 和non-transmitted allel 的个数,得到如下表格

TDT检验的统计量计算如下
(b - c)^ 2 / (b + c)这个统计量符合自由度为1的卡方分布。
plink 软件可以进行家系数据的GWAS分析,用法如下
plink --file mydata --tdt会生成plink.tdt文件,其中每列的含义如下
| Column | Meaning |
|---|---|
| CHR | Chromosome number |
| SNP | SNP identifier |
| A1 | Minor allele code |
| A2 | Major allele code |
| T | Transmitted minor allele count |
| U | Untransmitted allele count |
| OR | TDT odds ratio |
| CHISQ | TDT chi-square statistic |
| P | TDT asymptotic p-value |
| A:U_PAR | Parental discordance counts |
| CHISQ_PAR | Parental discordance statistic |
| P_PAR | Parental discordance asymptotic p-value |
| CHISQ_COM | Combined test statistic |
| P_COM | Combined test asymptotic p-value |
P值小于0.05的突变位点,就认为是与疾病相关的位点了。除了TDT检验外,plink还支持其他更多的检验方法。
看完上述内容,你们对基于家系数据的GWAS分析是怎样的有进一步的了解吗?如果还想了解更多知识或者相关内容,请关注亿速云行业资讯频道,感谢大家的支持。
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4580290/blog/4620997