今天就跟大家聊聊有关如何使用GenomeStudio 鉴定差异甲基化位点,可能很多人都不太了解,为了让大家更加了解,小编给大家总结了以下内容,希望大家根据这篇文章可以有所收获。
在对甲基化芯片进行差异分析之前,必须经过一个数据预处理的环节,预处理包括了归一化和背景降噪两个步骤,接下来看下GenomeStudio中进行差异分析下详细步骤。
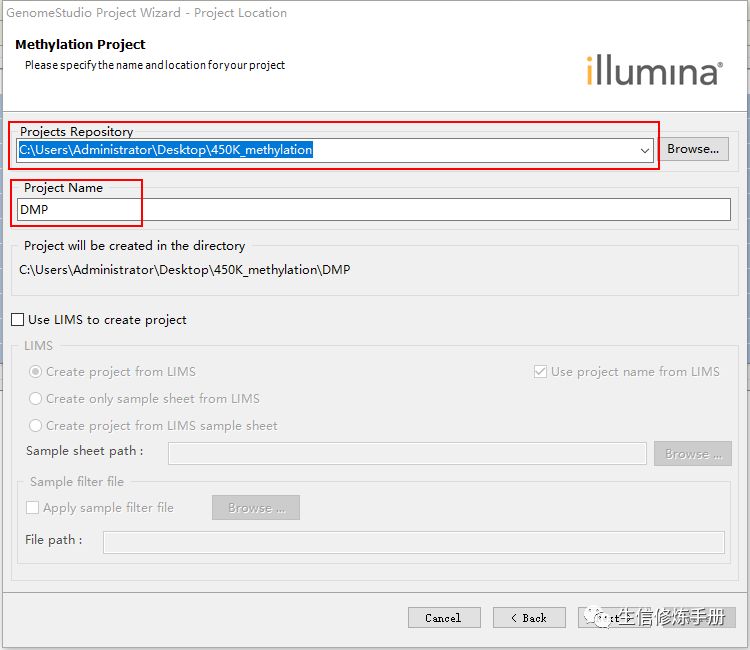
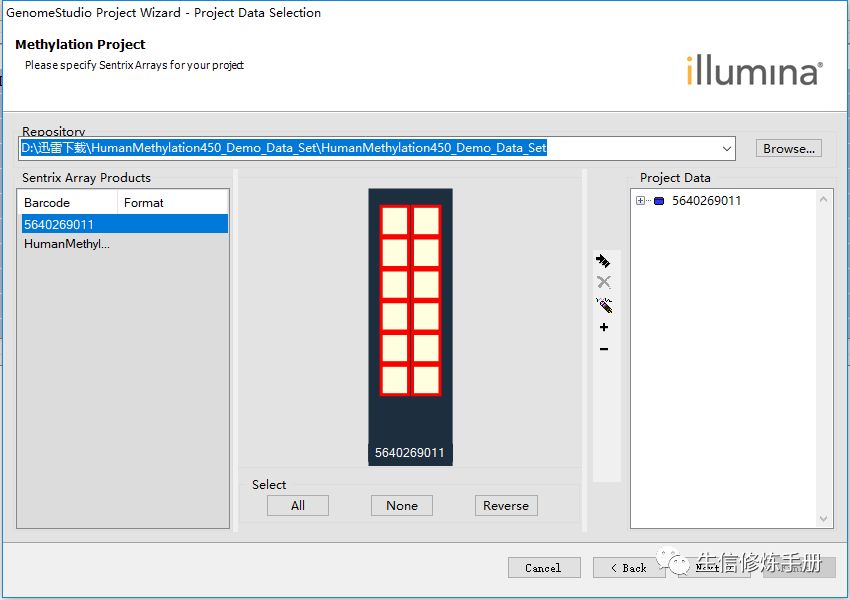
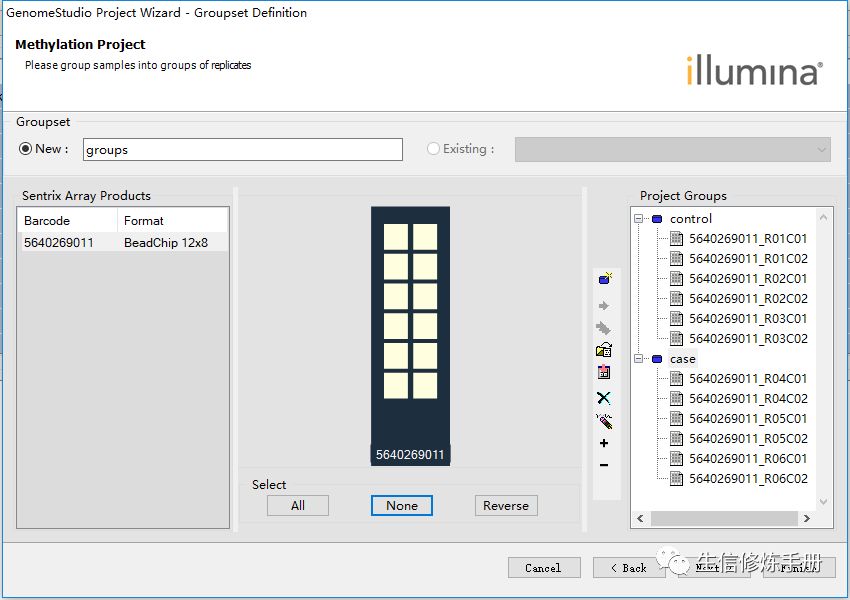
这里我随机选了6个样本作为control , 另外的6个样本作为case
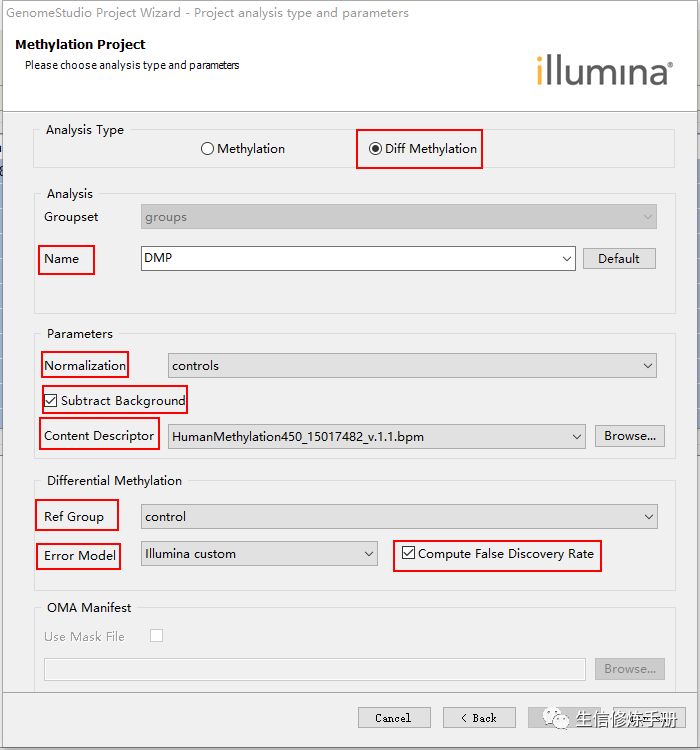
Analysis Type设置为Diff Methylation,
Name代表本次分析的名字
特别需要注意的是,必须进行归一化和背景降噪。Normalization 选择controls 算法, 勾选Subtract background前面的单选框
Content Descriptor 是芯片对应的bpm 格式的注释文件
Ref Group 选择对照组
Error Model 选择差异分析的方法,共有3种
illumina custom
Mann-Whitney
t-test
默认情况下,会有3个窗口的数据
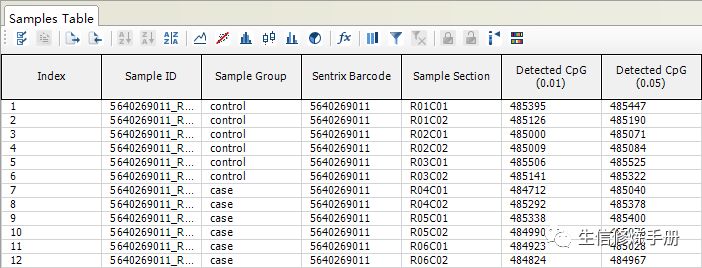
Sample Table
这个窗口包含了每个样本的基本信息,其中Detected CpG(0.01) 表示该样本中探针P值 < 0.01 的探针数
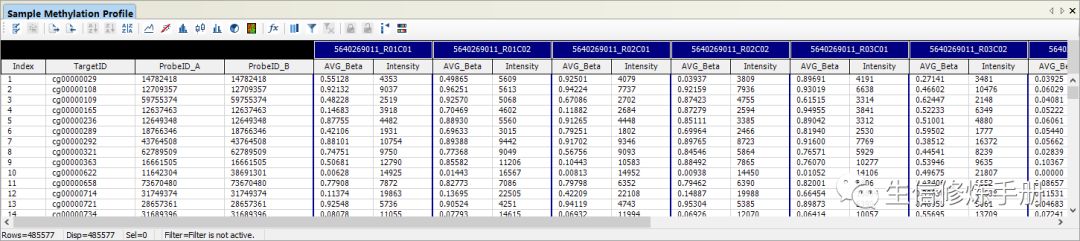
Sample Methylation Profile
样本的CpG表达谱, 在这个窗口中包含了CpG位点的表达值,比如Beta值,由于在导入数据时,进行了预处理,所以这里是预处理之后的表达量
Diff methylation
差异甲基化位点的结果
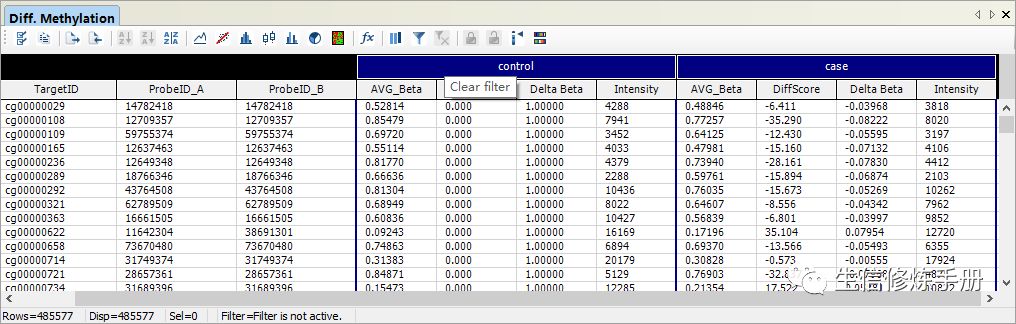
差异分析的结果中,p值时我们最常用的过滤手段,这里默认的结果中并没有给出P值,而是给出了1个DiffScore值。
DiffScore的计算公式如下:
DiffScore = 10 sgn(avg_case - avg_control) log10Pvalue
Diffscore 其实是和 Pvalue 有联系的,当
P= 0.05 时, DiffScore > 13 或者 DiffScore< -13
P = 0.01 时, DiffScore > 22 或者 DiffScore< -22
P = 0.001 时, DiffScore > 33 或者 DiffScore < -33
所以我们可以根据DiffScore值筛选差异分析的结果,如果你以P=0.05作为阈值, 那么DiffScore > 13 或者 DIffScore< -13 的探针就是差异探针了,而且DiffScore > 0 时是上调的,DiffScore <0 时是下调的。
对于下游的分析而言,我们肯定想要知道差异探针关联的基因是哪些,可以通过 按钮,在默认的结果中添加更多的列。
面板右边是一些隐藏的列,这里的列分为了Columns 和 SubColumns 两列,鼠标选中希望展示的列,然后单击中间的<=Show按钮,然后点击OK就可以了
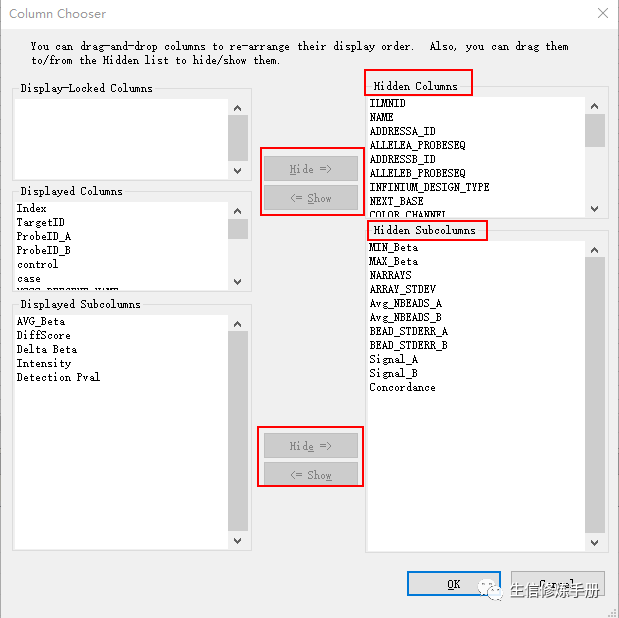
通常我们选择 UCSC开头的注释信息就够了,结果如下
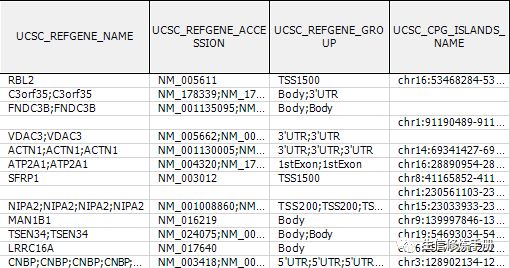
就可以看到每个探针对应的基因,然后对差异CpG位点对应的基因做功能富集分析。
看完上述内容,你们对如何使用GenomeStudio 鉴定差异甲基化位点有进一步的了解吗?如果还想了解更多知识或者相关内容,请关注亿速云行业资讯频道,感谢大家的支持。
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。