chip_seq质量评估中的PCA分析是怎样的,相信很多没有经验的人对此束手无策,为此本文总结了问题出现的原因和解决方法,通过这篇文章希望你能解决这个问题。
PCA我们称之为主成分分析,是一种经典的数据降维算法,通过将高维数据用几个主成分表示,从而将其映射到低维空间。在实际处理中,由于我们只能对二维和三维数据有直观的感受,所以通常绘制二维和三维的散点图。
PCA本质上属于排序分析的一种,降维之后的数据在二维或者三维平面通过散点图进行展示,两个样本点间的距离越接近,说明这两个样本越一致, PCA图在生物信息学中应用的非常广泛,该算法适用范围广泛,在基因组,转录组等多种数据分析中都有应用,本文主要介绍在chip_seq数据分析中的PCA分析。
在转录组中,我们可以通过基因表达谱来对样本进行PCA分析,在chip_seq数据分析中,为了得到类似基因表达谱的数据,研究人员提出了一种思想,将基因组划分为等长的区间,称之为bin,然后计算每个区间内的coverage。得到样本中所有bin的coverage之后,就可以利用该数据进行PCA分析。具体的操作步骤如下,通过deeptools来实现
输入文件为比对基因组产生的bam文件,用法示意如下
multiBamSummary bins \--bamfiles file1.bam file2.bam \--binSize 10000 \--numberOfProcessors 10 \--outRawCounts results.txt \-o results.npz \通过plotPCA命令实现,用法示意如下
plotPCA \-in results.npz \-o PCA.png
输出结果示意如下
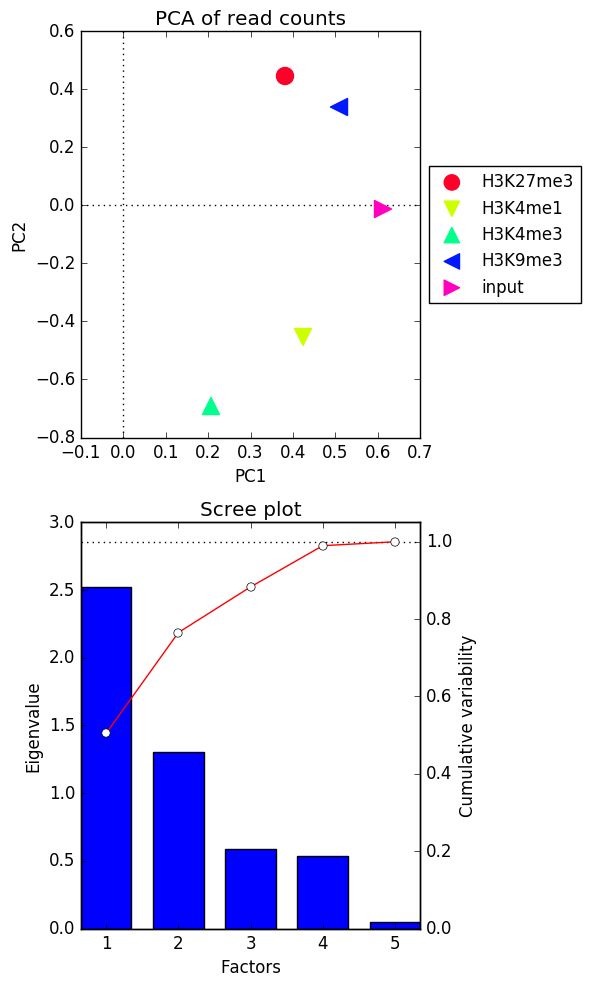
软件默认选择第一和第二主成分来绘制二维的散点图,在该图中通过观测样本点之间的距离,可以对数据质量做出一些基本判断,理论上讲,input和抗体处理的样本之间应该有较大距离,而生物学重复样本之间应该比较接近。
需要注意的是,前两个主成分的贡献率是一个比较重要的指标,假设两个主成分的贡献率之和为90%, 意味着二维散点图只能表征原始样本90%的信息,当贡献率太低时,散点图上表示的信息和原始样本的信息相去甚远,就不具有太大的参考意义了。
下半部分的Scree plot, 类似碎石图,只不过采用了双坐标轴的形式,蓝色柱状图表征了前5个主成分的特征值,红色曲线代表累计的特征值,每个点代表累计特征值的比例。当红色曲线趋于平缓时,说明即使再添加后面其他的主成分,所展示的信息也不会有显著变化了,即前几个主成分已经可以有效代表总体的信息了,在上图中,前4个主成分能够有效代表总体的信息。
虽然通过碎石图我们可以筛选出主成分,但是由于我们最多只能直观观察三维空间,所以PCA分析中最多只能绘制3维散点图,如果前3个主成分不能有效代表总体的信息,我们只能考虑使用其他降维算法了,这个问题也是所有降维算法的一个通病。
看完上述内容,你们掌握chip_seq质量评估中的PCA分析是怎样的的方法了吗?如果还想学到更多技能或想了解更多相关内容,欢迎关注亿速云行业资讯频道,感谢各位的阅读!
亿速云「云服务器」,即开即用、新一代英特尔至强铂金CPU、三副本存储NVMe SSD云盘,价格低至29元/月。点击查看>>
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。
原文链接:https://my.oschina.net/u/4580290/blog/4571247